Your new post is loading...

|
Scooped by
Life Sciences UPSaclay
January 4, 12:40 PM
|
Vous êtes chercheurs, étudiants, ingénieurs ou techniciens ? Vous travaillez dans le public ou le privé ? Vous avez besoin d’un accompagnement scientifique, technique et pluridisciplinaire dans les processus de découverte d’un médicament ? Alors, venez découvrir les plateformes technologiques dédiées à l’innovation thérapeutique de la Faculté de Pharmacie de l’Université Paris-Saclay, l’Unité Mixte de Services IPSIT, mardi 21 janvier de 13 H à 16 H. S’incrire : https://www.eventbrite.fr/e/billets-a-la-decouverte-des-plateformes-technologiques-de-lipsit-1040593552327 Le format retenu est résolument court, 1 heure de visite, sur inscription, suivie d’échanges autour d’une dégustation, afin de donner une vision globale de l’unité mixte de services et de rencontrer son personnel. Résolument à l’interface de la chimie, de la biologie et de la clinique, l’IPSIT établit des ponts entre la cible pathologique et le médicament. Le processus de découverte d’un médicament requiert une approche multidisciplinaire associant des savoir-faire complémentaires, notamment dans les domaines de la chimie, de la galénique, de la biologie structurale, de la biologie moléculaire et cellulaire, sans oublier la génétique et la pharmacologie. L’IPSIT, par ses plateformes technologiques, apporte aux équipes une aide scientifique essentielle dans ce processus. L’IPSIT participe aussi à l’animation scientifique et à la formation des étudiants et des personnels tout en contribuant au rapprochement d’équipes d’horizons différents et à la transdisciplinarité des collaborations. Découvrez ainsi : - le pôle OMICS avec ses plateformes de transcriptomique et génomique (ACTAGen), protéomique (PROTEOMIC), spectrométrie de masse et lipidomique (SAMM)
- le pôle Imagerie Cellulaire avec ses plateformes de microscopie photonique super-résolue et d’analyse d’images (MIPSIT), de cytométrie en flux (CYM) et d’histopathologie (PHIC)
- le pôle Interaction avec la plateforme de criblage moléculaire (CIBLOT)
- Ainsi que des services transverses constituées par une animalerie ultra-moderne équipée de dispositifs d’exploration fonctionnelle (ANIMEX), une verrerie scientifique et technique (VERRE SCIEN-TECH) et un plateau de bio-informatique (BIOINFO).
-> Contact : Valerie Domergue (valerie.domergue@universite-paris-saclay.fr), Directrice de l’IPSIT A propos de l’IPSIT. IPSIT (Ingénierie et Plateformes au Service de l’Innovation Thérapeutique) est une Unité Mixte de Service placée sous les tutelles conjointes de l’UPSaclay (UMS-IPSIT), l’Inserm (US31) et le CNRS (UAR3679). L’IPSIT regroupe 10 plateformes techniques, organisées en trois pôles technologiques (IMCELLF, OMICS et INTERACTIONS) et trois plateformes transverses. L’IPSIT se veut résolument à l’interface de la chimie, de la biologie et de la clinique en établissant le lien entre la cible pathologique et le médicament. L’IPSIT est adossée à une Structure Fédérative de Recherche (SFR) qui rassemble l’UMS et 25 équipes de recherche. Enfin, l’IPSIT participe à l’animation scientifique et à la formation des étudiants et des personnels tout en contribuant au rapprochement d’équipes d’horizons différents et à la transdisciplinarité des collaborations. Voir aussi leur FOCUS PLATEFORME décrivant toutes leurs expertises !

|
Scooped by
Life Sciences UPSaclay
January 8, 5:58 PM
|
Fonction effectrice des portions Fc des anticorps monoclonaux dirigés contre les points de contrôle immunitaire en oncologie
Dans une revue publiée dans Immunological Reviews, les chercheurs du Laboratoire de Recherche Translationnelle en Immunothérapie (UMR-S 1015 INSERM/UPSaclay/Gustave Roussy, Villejuif) font le point sur les anticorps monoclonaux antagonistes (mAbs) ciblant les points de contrôle immunitaires inhibiteurs qui ont révolutionné le domaine de l'oncologie. En effet, CTLA-4, PD-1 et LAG3 sont trois protéines transmembranaires qui sont exprimées par des sous-ensembles de lymphocytes T et qui jouent un rôle dans la régulation des réponses immunitaires adaptatives. Le blocage de ces points de contrôle immunitaire (ou de leurs ligands) à l'aide d'anticorps antagonistes peut entraîner des régressions tumorales et des rémissions durables chez certains patients atteints de cancer. Leur mécanisme d'action, leur efficacité et leurs effets secondaires associés sont liés à leur affinité avec les récepteurs Fc gamma (FcγR) (appelés « fonctions effectrices »). Certains de ces anticorps ont des isotypes ayant une forte affinité pour l'activation des FcγR et peuvent donc induire une ADCC/ADCP (cytotoxicité à médiation cellulaire dépendante des anticorps) et conduire à la mort des lymphocytes. D’autres anticorps ont une faible affinité et n’engendrent donc pas ces réactions associées. Enfin certains anticorps ont même été mutés dans le but d’obtenir une faible affinité pour les FcγR afin de limiter les phénomènes d’ADCC/ADCP et donc les effets indésirables. Les chercheurs passent en revue ici les douze anticorps monoclonaux dirigés contre les points de contrôle immunitaires approuvés par la FDA et l'EMA et discutent de l'impact de leur fonction effectrice Fc sur leur mode d’action. -> Contact : aurelien.marabelle@gustaveroussy.fr / romane.martineau@gustaveroussy.fr

|
Scooped by
Life Sciences UPSaclay
January 8, 5:15 PM
|
Le directeur du laboratoire antidopage français Magnus Ericsson revient pour « L'Équipe » sur le défi rempli pendant les JO de Paris et sur le futur de la lutte antidopage. Les spectromètres de masse et autres analyseurs d'hématologie sont encore présents en nombre en ce début décembre. Pour répondre aux cadences de contrôle des Jeux Olympiques et Paralympiques de Paris, le laboratoire antidopage français (LADF) avait accueilli, depuis son déménagement de Châtenay-Malabry (Hauts-de-Seine) à Orsay (Essonne) en juin 2023, une multitude de machines pour satisfaire au rythme infernal des contrôles pendant cette période. Certaines sont déjà reparties vers divers labos, d'autres attendent d'être vendues et un gros contingent continue de tourner puisque les contrôles antidopage ne se sont évidemment pas arrêtés à l'extinction de la vasque. C'est dans cette atmosphère de travail studieuse et tranquille que Magnus Ericsson, le patron des lieux, nous a accueillis pour revenir sur un moment inédit de l'histoire du labo français. Lire la suite de l’article dans l’Equipe -> Contact : m.ericsson-ladf@universite-paris-saclay.fr

|
Scooped by
Life Sciences UPSaclay
January 8, 3:58 PM
|
Tous les êtres vivants actuels ont un ancêtre commun, une petite cellule que la communauté scientifique a baptisée LUCA. De quelle nature est-il ? A quand peut-on le dater ? Quel a été son impact sur son environnement ? Avec - Guillaume Lecointre, Professeur au Museum, National d'Histoire Naturelle (MNHN). Chercheur en systématique
- Purificación López-García, Biologiste, directrice de recherche CNRS au laboratoire Ecologie Systématique Evolution –ESE (CNRS/AgroParisTech/UPSaclay, Gif-sur-Yvette)
Son nom est LUCA, acronyme anglais pour parler du dernier ancêtre commun universel à tous les organismes vivants. Il est au cœur de l’une des enquêtes les plus fascinantes de la biologie. On en parle aujourd’hui. Depuis le développement de la biologie moléculaire au XXe siècle, on sait que tous les êtres vivants partagent un même code génétique permettant de transmettre les informations entre les molécules qui le composent. Cette unité dans la grande diversité du vivant, suggère l’existence lointaine d’un ancêtre commun réunissant toutes ces caractéristiques. Pourtant, il est encore difficile de dessiner le portrait de ce premier ancêtre. Quel âge a-t-il ? De quoi était-il composé ? Et dans quel environnement s’est-il développé ? On tente une ébauche de réponse. Pour écouter ou réécouter le podcast de l’émission du 7 janvier 2025, cliquez ICI. -> Contact : puri.lopez@universite-paris-saclay.fr
Merci à toutes et tous pour vos nombreuses contributions ! Excellente année 2025 avec toujours plus de belles découvertes dans le domaine des Sciences de la Vie !

|
Scooped by
Life Sciences UPSaclay
December 28, 2024 12:13 PM
|
L'EMT (ou transition épithélio-mésenchymateuse) est un processus biologique par lequel une cellule perd ses caractéristiques usuelles pour acquérir des propriétés lui permettant de se déplacer. L’EMT est impliquée dans la formation de l’embryon ou dans la réparation des tissus chez l’adulte. Mais lorsqu’elle est altérée, l’EMT contribue à la formation de fibrose ou dans le cas d’une cellule tumorale, à former des métastases. Dans les cancers, ce changement est comparable à une métamorphose cellulaire qui permet aux cellules cancéreuses de devenir plus agressives et résistantes aux traitements. L'EMT est donc un acteur clé dans la progression tumorale. L’EMT est extrêmement étudiée au niveau moléculaire et cellulaire car une meilleure compréhension de ce processus permettra le développement de nouvelles approches thérapeutiques. Cependant les changements traductionnels qui se produisent durant l’EMT n’ont été jusqu’à présent que très peu étudiés. Dans une étude parue dans PNAS, les scientifiques de l’équipe Genomics, Structure and Translation de l’I2BC (CNRS/CEA/UPSaclay, Gif-sur-Yvette) ont identifié que la surexpression d’une seule protéine ribosomique, appelée RPL36A, est suffisante pour induire l’EMT. Ils ont aussi mis en évidence les différents changements traductionnels qui se produisent durant l’EMT. Ce travail révèle l’importance des remaniements du ribosome lui-même permettant de moduler finement la traduction lors du changement d’identité cellulaire qui se produit durant l’EMT. Lire aussi l'Actu CNRS -> Contact : olivier.namy@i2bc.paris-saclay.fr

|
Scooped by
Life Sciences UPSaclay
December 28, 2024 4:21 PM
|
Comment évaluer la vulnérabilité des vertébrés terrestres face aux invasions biologiques ?
Les espèces exotiques envahissantes (EEE) constituent une menace majeure pour la biodiversité. Afin de réduire les introductions et des impacts des EEE, il est essentiel d’identifier les zones qui y sont les plus vulnérables pour prioriser les actions de prévention pour préserver la biodiversité. Une équipe scientifique du laboratoire Ecologie Systématique Evolution –ESE (CNRS/AgroParisTech/UPSaclay, Gif-sur-Yvette), du Muséum national d’Histoire naturelle et de l’Université d’Edimbourg (Ecosse) a cartographié la vulnérabilité mondiale aux invasions biologiques. Leurs travaux ont été publiés dans la revue Global Change Biology. En analysant la répartition spatiale de plus de 300 EEE et la sensibilité de plus de 1 600 espèces de vertébrés (oiseaux, mammifères et reptiles) à ces invasions, cette étude a mis en évidence que 38% des terres mondiales sont exposées aux invasions biologiques, mais que l’exposition seule ne suffit pas pour évaluer les risques. Par exemple, les oiseaux sont particulièrement vulnérables dans les zones côtières d’Amérique du Nord et du Sud, d’Afrique du Sud, d’Australie, et d’Asie du Nord-Est, ainsi que dans les îles océaniques, en Nouvelle-Zélande, et certaines régions européennes. Les régions de très faible vulnérabilité (faible sensibilité et faible exposition aux espèces non-natives envahissantes) incluent les latitudes polaires, l’Amazonie, la majeure partie de l’Afrique, et l’Asie centrale. Toutefois, ces zones correspondent souvent à des lacunes de données, laissant entrevoir des zones potentiellement vulnérables encore non identifiées. Ce travail souligne les besoins urgents de connaissances pour protéger les espèces natives et leurs habitats, en particulier dans les régions insulaires et côtières à haute biodiversité. Mais surprise : des zones géographiques supposément bien étudiées comme l’Europe de l’Ouest étaient aussi dépourvues de données de qualité. Bien que les savoirs sur la biodiversité augmentent, les connaissances sur les impacts des EEE demeurent insuffisantes pour évaluer la menace qu’elles représentent dans de nombreuses zones du monde. -> Contact : clara.marino@universite-paris-saclay.fr / celine.bellard@universite-paris-saclay.fr

|
Scooped by
Life Sciences UPSaclay
December 28, 2024 4:40 PM
|
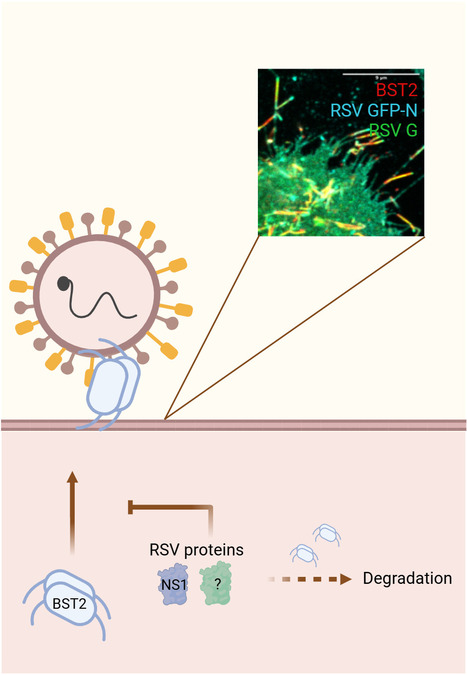
Dans une étude publiée dans PLoS Pathogens, l’équipe « Mécanismes moléculaires de la multiplication des Pneumovirus » (UVSQ/UPSaclay/INSERM/Institut Pasteur/Paris Cité), en collaboration avec une équipe de l’institut Cochin, démontre pour la première fois que la protéine cellulaire BST2 se comporte comme un facteur de restriction à l’infection par le virus respiratoire syncytial (RSV). Le RSV est le principal agent viral responsable d'infection sévère des voies respiratoires inférieures chez les enfants, avec plus de 30 millions de cas dans le monde chaque année. La réponse immunitaire innée est un déterminant majeur de la gravité de la maladie. Les auteurs démontrent que l’inhibition de BST2, une protéine clé de la réponse immunitaire innée, entraîne une augmentation significative de la production virale au cours d'une infection en multiples cycles, ce qui suggère un rôle inhibiteur au cours des dernières étapes du cycle de multiplication du RSV. Katherine Marougka (unité Infection & Inflammation – 2I UMR-S 1173 Inserm/UPSaclay/UVSQ, Montigny-le-Bretonneux) et ses collègues mettent en évidence que le virus contrecarre cet effet en diminuant la quantité de BST2 à la surface des cellules. BST2 se distribuant avec les particules virales du RSV à la membrane plasmique, ils proposent que BST2 se comporte comme un facteur de restriction en attachant les particules du RSV à la surface des cellules infectées. Les auteurs démontrent également que la protéine NS1 du RSV interagit avec BST2 et participe au mécanisme par lequel le RSV régule BST2. La régulation de BST2 dans des cellules proches de ses cibles naturelles souligne la pertinence de ces données et pourrait ouvrir la voie à de nouvelles stratégies de contrôle de l'infection. -> Contact : delphine.sitterlin@uvsq.fr

|
Scooped by
Life Sciences UPSaclay
December 28, 2024 4:58 PM
|
Caractéristiques cliniques et moléculaires des patients jeunes (<50 ans) avec un cancer du pancréas
L’adénocarcinome du pancréas (PDAC) représente un défi majeur en santé publique et pourrait devenir la deuxième cause de mortalité par cancer dans les pays développés. L’incidence des cancers pancréatiques précoces (EOPC), diagnostiqués avant 50 ans, est en augmentation. Une étude portant sur 878 patients suivis à Gustave Roussy, dont 113 EOPC, a été menée par Alice Boilève (UMR-S 1279 INSERM/UPSaclay/Gustave Roussy, Villejuif) et ses collaborateurs, dont les résultats sont publiés dans International Journal of Cancer. Les EOPC présentent un état général comparable à celui des patients âgés de plus de 50 ans (nEOPC), mais sont plus souvent diagnostiqués à un stade métastatique (70% contre 58,3%), avec une prédominance de métastases hépatiques (60,2% contre 43,9%). La survie globale médiane était similaire entre les deux groupes (18,1 mois). Chez les patients opérés, la survie sans récidive était équivalente. Parmi les patients métastatiques, la survie sans progression après une première ligne de traitement était comparable, bien que les EOPC aient reçu davantage de lignes thérapeutiques (≥2 lignes pour 72,3% contre 58,1%). Sur le plan moléculaire, les EOPC présentaient une charge mutationnelle tumorale (TMB) plus faible (1,42 contre 2,95 mutations/Mb) et moins d’altérations de CDKN2A/B, tandis que les mutations de KRAS et BRCA1/2 étaient similaires. Près de 19% des patients avaient des altérations moléculaires exploitables, et 31 ont bénéficié de traitements ciblés. Enfin, malgré une agressivité clinique marquée, les EOPC étaient moins souvent associés à un phénotype basal-like. En conclusion, les EOPC se distinguent par un diagnostic plus tardif, des caractéristiques moléculaires spécifiques et la nécessité d’une prise en charge adaptée. -> Contact : alice.boileve@gustaveroussy.fr

|
Scooped by
Life Sciences UPSaclay
December 28, 2024 5:18 PM
|
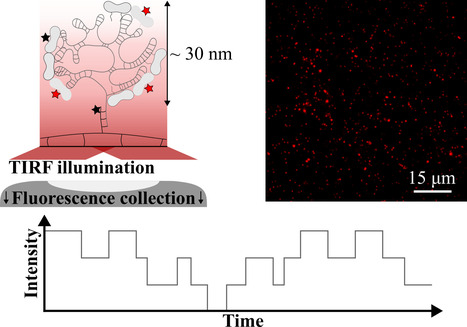
Une des étapes cruciales du cycle de vie d’un virus met en œuvre un processus complexe d’auto-assemblage malgré l’encombrement macromoléculaire et l’activité enzymatique intense régnant dans le cytoplasme. Les virus sphériques les plus simples co-assemblent leur capside - c’est à dire leur coque protéique - avec leur génome souvent à ARN simple brin. Si plusieurs techniques de moyenne d’ensemble permettent d’élucider certains mécanismes clés de l’assemblage grâce à leur excellente résolution spatio-temporelle, elles échouent à rendre compte de la variabilité statistique d’un site d’assemblage à l’autre. Dans un étude publiée dans Nano Letters, des chercheurs du Laboratoire Lumière, Matière et Interface – LuMIn (UPSaclay/ENS Paris-Saclay/CNRS/CentraleSupélec, Gif-sur-Yvette) et du Laboratoire de Physique des Solides (UPSaclay/CNRS, Orsay) ont implémenté une méthode expérimentale pour sonder la dynamique d’assemblage à l’échelle des protéines individuelles. Pour ce faire, ils ont utilisé la microscopie de fluorescence à réflexion totale interne (TIRF) et ont suivi l’évolution de l’intensité lumineuse de centaines de sites d’assemblage simultanément à mesure que des protéines fluorescentes s’y accrochent ou au contraire s’en détachent (Figure). Grâce à un algorithme de détection de marches combiné à des analyses statistiques, ils ont estimé des grandeurs microscopiques telles que le taux d’accrochage et le temps de résidence moyen. Ils ont de plus déterminé un jeu de constantes de vitesse modélisant la cinétique d’assemblage à partir de mesures hors équilibre. L’imagerie de fluorescence en molécule unique se révélera cruciale pour élucider l’auto-assemblage de virus à l’échelle moléculaire, particulièrement dans des milieux encombrés et actifs mimant les conditions cytoplasmiques. Légende Figure : Principe de la méthode expérimentale reposant sur la microscopie TIRF. A mesure que les protéines fluorescentes de la capside s’accrochent ou se détachent des sites d’assemblage, l’intensité lumineuse de chacun de ces sites varie au cours du temps reflétant la dynamique des évènements. -> Contact : guillaume.tresset@universite-paris-saclay.fr /karen.perronet@ens-paris-saclay.fr

|
Scooped by
Life Sciences UPSaclay
January 4, 11:53 AM
|
Médecin Néphrologue et ancienne Chef de Clinique des Hôpitaux de Paris, ayant effectué son post-doctorat au sein du Karolinska Institutet de Stockholm, Anne-Laure Faucon travaille dans l’équipe « Epidémiologie Clinique » du Centre de Recherche en Epidémiologie et Santé des Populations – CESP (UMR-S 1018 INSERM/UVSQ/UPSaclay, Villejuif), spécialisée en épidémiologie-biostatistiques appliquées aux maladies rénales. Ses travaux ont porté sur l’étude de la volémie chez les patients suivis pour une maladie rénale chronique. Elle a montré que cette volémie augmente au cours de l’évolution et est associée au non-contrôle tensionnel avec un risque de décès. Elle a développé un algorithme mathématique pouradapter la posologie du traitement diurétique.

|
Scooped by
Life Sciences UPSaclay
December 29, 2024 4:42 AM
|
Le Professeur Daniele De Luca a été élu membre honoraire de l’American Pediatric Society, la plus ancienne académie scientifique de pédiatrie fondée en 1888. Cette nomination, réservée aux non-américains, n’est que rarement attribuée. Le Pr De Luca est l’unique Européen de cette promotion et vraisemblablement le premier Français. Le Pr Daniele De Luca est Chef du Service de Réanimation Néonatale du Groupe Hospitalier APHP/UPSaclay, Hôpital Antoine Béclère depuis 2013 et PUPH à l’Université Paris-Saclay de Pédiatrie depuis 2021, et membre de l’UMR-S 999 « Hypertension pulmonaire : physiopathologie et innovation thérapeutique » (INSERM/UPSaclay). Il est actuellement “Immediate Past President" de la European Society for Paediatric and Neonatal Intensive Care (ESPNIC), la plus grande société scientifique pédiatrique européenne qui regroupe les réanimateurs néonatales et pédiatriques du continent. Son groupe est spécialisé dans la réanimation des nouveau-nés et des nourrissons en danger de mort (prématurité, SDRA, bronchiolite, cardiopathie congénitale, insuffisance rénale, hypothermie thérapeutique). Pendant la pandémie, il a été activement impliqué et a découvert la transmission transplacentaire du SRAS-CoV-2 (Nature Communications 2020, travail cité plus de 700 fois) et pour cette raison, il a été nommé consultant auprès de l'Organisation mondiale de la santé (OMS). Auteur d'environ 500 ouvrages scientifiques et de dizaines d'ouvrages et chapitres (H-index 52). Il a reçu le prix européen du jeune chercheur ESPR en 2010 et son groupe a remporté le prix Bengt Robertson en 2018. Il a également reçu de l’APHP le Trophée APPINNOV 2023 pour l’innovation médicale et notamment pour le brevet de l’échographie pulmonaire quantitative pour le monitorage et prédiction de la dysplasie broncho-pulmonaire du prématuré. Il est président honoraire de la branche de médecine pédiatrique de l'Association de santé Asie-Pacifique. Il est Editeur du European Journal of Pediatrics, la plus ancienne revue scientifique pédiatrique du continent. Nommé Chevalier de la République Italienne en 2021. -> Contact : daniele.deluca@aphp.fr

|
Scooped by
Life Sciences UPSaclay
December 20, 2024 9:11 AM
|
Purificación López-García, élue membre de l’Académie des Sciences
Purificación López-García, directrice de recherche CNRS au laboratoire Ecologie Systématique Evolution –ESE (CNRS/AgroParisTech/UPSaclay, Gif-sur-Yvette) a été élue membre de l’Académie des Sciences. Purificación López-García bénéficie d’une excellente reconnaissance nationale et internationale pour ses contributions remarquables à l’identification des formes de vie dans les environnements naturels les plus inhospitaliers, qui lui ont permis d’être l’une des grandes spécialistes en biologie de l’évolution, des origines de l’arbre du vivant. Les résultats des élections qui viennent d’avoir lieu devront prochainement être ratifiés par décret officiel du président de la République. Une cérémonie solennelle de réception des nouveaux membres se tiendra ensuite sous la Coupole de l’Institut de France le mardi 3 juin 2025, de 15h à 17h30, au son des tambours de la Garde républicaine. Retrouver le communiqué complet de l’Académie des sciences
|

|
Scooped by
Life Sciences UPSaclay
January 8, 6:08 PM
|
Construire un médicament à l’intérieur de cellules cancéreuses

|
Scooped by
Life Sciences UPSaclay
January 8, 5:46 PM
|
Les maladies à prions, ou encéphalopathies spongiformes transmissibles (EST), sont des pathologies neurodégénératives provoquées par l'accumulation de conformères mal repliés (PrPSc) de la protéine prion cellulaire (PrPC). Ces conformères pathologiques se propagent dans le système nerveux central, transformant PrPC en assemblages insolubles. Les variations cliniques des EST, attribuées à des souches spécifiques de prions, résultent de différences dans les structures de PrPSc. Dans une étude publiée dans iScience, des chercheurs de l’équipe Macro-Assemblages Protéiques et Maladies à Prions (MAP2) de l’unité Virologie et Immunologie Moléculaires – VIM (UMR 0892 INRAE/UVSQ/UPSaclay, Jouy-en-Josas) ont développé un modèle stochastique de réaction-diffusion basé sur l’algorithme de Gillespie pour explorer la neuro-invasion des prions. Ce modèle intègre des avancées récentes sur la diversité structurelle de PrPSc et les réponses des tissus à l’accumulation de PrPSc. Les résultats montrent que la combinaison de non-linéarités dans ce système complexe reproduit des caractéristiques clés de la propagation des prions, comme les phénotypes spécifiques des souches, les tropismes tissulaires, et la co-propagation de plusieurs souches. De plus, le modèle clarifie le rôle du connectome dans le processus de neuro-invasion. Cette approche propose une alternative innovante aux paradigmes actuels pour comprendre les mécanismes des maladies à prions. Elle ouvre la voie à des perspectives prometteuses pour étudier les interactions entre souches et tissus, tout en mettant en lumière l’importance de la modélisation pour décrypter les dynamiques de ces pathologies complexes. -> Contact : basile.fornara@inrae.fr / human.rezaei@inrae.fr

|
Scooped by
Life Sciences UPSaclay
January 8, 5:01 PM
|
Portrait Jeune Chercheur – Florent Peglion, Chercheur en biologie cellulaire du cancer
Florent Peglion est Chargé de Recherche INSERM en biologie cellulaire, actuellement en poste dans l’unité Dynamique des Cellules Tumorales UMR-S 1279 (INSERM/UPSaclay/Gustave Roussy, Villejuif), au sein de l’équipe Biologie des Cancers Digestifs Avancés. Intéressé par la façon dont les cellules se polarisent et migrent dans des conditions normales et pathologiques, Florent cherche spécifiquement à comprendre comment les cellules au sein d’un amas cellulaire en mouvement (cluster) coordonnent leur réponse aux stimuli extérieurs pour favoriser la migration du groupe. Ses recherches sont appliquées à la dissémination tumorale collective à l’œuvre pendant la progression métastatique des cancers colorectaux et pancréatiques. Après une formation d’ingénieur agronome, Florent effectue une thèse en biologie cellulaire à l’Institut Pasteur sur la dynamique des jonctions intercellulaires pendant la migration cellulaire collective. S’en suivent cinq années de postdoctorat au Francis Crick Institute à Londres pour étudier les mécanismes permettant aux cellules de se polariser afin d’effectuer une division cellulaire asymétrique puis cinq autres années de nouveau à l’Institut Pasteur pour comprendre l’impact de la perte de la polarité cellulaire dans l’invasion des tumeurs du cerveau malignes (glioblastomes). Au cours de ces deux postdoctorats, il travaille sur les organismes modèles C.elegans et développe un essai de greffe orthotopique d’échantillons de glioblastomes de patients dans le cerveau des larves de poisson-zèbre pour y observer en temps réel leur mode d’invasion. En 2023, souhaitant se rapprocher davantage de la clinique, il rejoint l’équipe Invasion Collective de l’U1279 (nouvellement nommée Biologie des Cancers Digestifs) à Gustave Roussy et y obtient un poste de CR à l’INSERM (section CSS2) l’année suivante. A partir d’échantillons de patients atteints de cancer colorectaux métastatiques Florent cherche à mettre en évidence les déterminants moléculaires d’un nouveau mode d’invasion tumorale utilisé par une partie de ces cancers pour disséminer : l’invasion collective dite amiboïde. Florent voue un grand intérêt à la transmission au plus grand nombre des travaux de recherche en s’impliquant dans divers projets de vulgarisation de la recherche. Il a travaillé aussi à lutter contre la mésinformation scientifique en collaborant pour le site https://science.feedback.org/fr/ qui s’associe aux réseaux sociaux pour alerter sur la dangerosité de certains contenus pseudo-scientifiques. Enfin, Florent donne des séminaires sur la dissémination tumorale dans le cadre de différents UE et DU (Sorbonne Université, Université Paris-Cité). « L'art et la science, c'est tâcher de comprendre. L'échec et la réussite sont tout à fait secondaires. » - Alberto Giacometti -> Contact : florent.peglion@gustaveroussy.fr

|
Scooped by
Life Sciences UPSaclay
January 6, 11:25 AM
|
Sylvie Retailleau, nouvelle membre du Conseil de surveillance de l’Institut Curie
Sylvie Retailleau, ancienne ministre de l’Enseignement supérieur et de la Recherche, intègre le Conseil de surveillance de l’Institut Curie. Instance pilier de la gouvernance de l’Institut Curie, le Conseil de surveillance a pour missions principales de contrôler et de s’assurer de la bonne gestion du Directoire. Présidé par Daniel Thierry et composé de 18 membres, il est assisté, dans ses missions de surveillance, par trois comités ad hoc : le Comité des rémunérations, le Comité d’audit et des finances et le Comité d’éthique. Sylvie Retailleau a été nommée membre du Conseil de surveillance lors de sa séance du 12 décembre 2024. « Rejoindre le Conseil de surveillance de l’Institut Curie représente pour moi une opportunité de m’investir au cœur de cette fondation d’excellence, en soutenant ses missions fondamentales de recherche, de soin et de transmission des savoirs. J’aurai à cœur d’apporter une vision stratégique et collaborative pour accompagner les ambitions de l’Institut Curie dans la lutte contre le cancer », déclare Sylvie Retailleau, nouveau membre du Conseil de surveillance de l’Institut Curie. Professeure des universités en électronique à l'Université Paris-Saclay, Sylvie Retailleau a été ministre de l’Enseignement supérieur et de la Recherche de mai 2022 à septembre 2024, après avoir présidé l’Université Paris-Saclay de mars 2020 à mai 2022 et la ComUE « Université Paris-Saclay » en 2019. Elle est également membre de plusieurs conseils d’administration d’institutions majeures, telles que le CNRS, l’INRIA, le CEA et l’ENS Paris-Saclay. Son domaine de recherche porte sur la modélisation physique, la simulation de circuits et le développement d’architectures innovantes pour les générations futures de composants intégrés. Sylvie Retailleau a été décorée chevalier de la Légion d’honneur et officier de l’ordre national du Mérite. Lire le communiqué de presse de l'Institut Curie

|
Scooped by
Life Sciences UPSaclay
December 28, 2024 11:55 AM
|
La biologie synthétique permet le calcul biologique, grâce à laquelle les systèmes biologiques modifiés répondent aux entrées de manière programmée. Cependant, le calcul biologique traditionnel des systèmes unicellulaires est limité par des contraintes de ressources, qui limitent la quantité d'ADN étranger pouvant être exprimé dans une cellule. Pour surmonter ce problème, l'informatique biologique « distribuée » utilise des communautés de cellules pour diviser les tâches informatiques de grande envergure. Cette approche nécessite cependant une communication efficace entre les cellules, ce qui est difficile en raison du nombre limité de molécules de signalisation uniques (orthogonales). Dans une recherche publiée dans Nucleic Acids Research, des scientifiques de l’Institut MICALIS (Inrae/AgroParisTech/UPSaclay, Jouy-en-Josas) et de l'ENS Paris-Saclay (Laboratoire Méthodes Formelles, Gif-sur-Yvette), rassemblés au sein d’un Cellular Computing Group, ont relevé ce défi en créant un nouveau système de communication utilisant des bactériophages modifiés pour transférer des messages ADN codant des gènes d'ARN guide entre les cellules bactériennes. Cette méthode, appelée « CRISPRi intercellulaire » (i-CRISPRi), combine le transfert d'ADN avec l'interférence CRISPR (CRISPRi), permettant de contrôler les cellules expéditrices sur l'expression des gènes dans les cellules réceptrices. Les messages ADN programmables permettent divers signaux qui facilitent une bande passante élevée et une communication rapide. Les chercheurs ont validé la méthode i-CRISPRi en construisant huit signaux ADN uniques et en concevant des ensembles bactériens capables d'exécuter des portes logiques numériques telles que NOT, YES, AND et AND-AND-NOT. Ces portes logiques impliquaient jusqu'à sept types bactériens différents, démontrant la capacité d'effectuer des tâches de calcul distribuées. Les principales conclusions ont révélé que l'efficacité de la communication dépend des phases de croissance et de la concurrence des ressources entre les bactéries. Cette approche améliore la puissance de calcul des communautés bactériennes, leur permettant d'effectuer des tâches complexes et distribuées. Elle fournit également une base pour la construction de circuits biologiques plus grands et plus complexes, semblables à des réseaux informatiques interconnectés, faisant progresser le domaine de la biologie synthétique et ses applications.
-> Contact : mfuegger@lmf.cnrs.fr / thomas@thomasnowak.net / manish.kushwaha@inrae.fr

|
Scooped by
Life Sciences UPSaclay
December 26, 2024 5:58 PM
|
Une étude franco-internationale, publiée dans la revue américaine Stroke, révèle que travailler plus de dix heures par jour durant au moins cinquante jours par an pourrait augmenter de 29 % le risque d’accident vasculaire cérébral (AVC). Cette recherche, menée conjointement par des chercheurs de l’hôpital Raymond-Poincaré (AP-HP), de l’UVSQ/Université Paris-Saclay et de l’Inserm (UMR 1168/UMS 11, Villejuif), avec la collaboration d’experts américains, européens et japonais, s’appuie sur les données de la cohorte française Constances. Cette dernière regroupe 200 000 adultes âgés de 18 à 69 ans, suivis dans les Centres d’examens de santé de la Sécurité sociale. Les résultats montrent une corrélation encore plus marquée pour les individus exposés à ces conditions de travail pendant plus de dix ans, avec un risque d’AVC doublé. L’étude a également identifié des différences significatives selon les catégories professionnelles et l’âge, tout en prenant en compte des facteurs de risque comme l’hypertension, le diabète et le tabagisme. Ces découvertes soulignent l’importance d’adopter des horaires de travail flexibles et raisonnables pour prévenir les AVC, mettant en évidence la nécessité de stratégies de prévention ciblées pour les travailleurs à haut risque. Lire la suite de l’article Doctissimo -> Contact : alexis.descatha@inserm.fr

|
Scooped by
Life Sciences UPSaclay
December 28, 2024 4:31 PM
|
Clostridioides difficile est une cause majeure d'infections nosocomiales associées à l'antibiothérapie, classée comme une menace urgente de résistance aux antibiotiques. Ce pathogène interagit avec l'hôte et les communautés microbiennes intestinales au cours de l'infection, mais les mécanismes de ces interactions restent largement à découvrir. Les ARN non codants (ARNnc) contribuent à la virulence bactérienne et aux réponses de l'hôte, mais leur expression n'a pas été étudiée au cours de l'infection par C. difficile. Dans une étude publiée dans mSystems, un travail collaboratif des laboratoires de l’I2BC (CNRS/CES/UPSaclay, Gif-sur-Yvette) et l’Institut MICALIS (Inrae/AgroParisTech/UPSaclay, Jouy-en-Josas) a tiré parti du modèle murin conventionnel de l'infection par C. difficile pour étudier simultanément la dynamique de l'expression des gènes chez le pathogène, son hôte et la composition du microbiote intestinal, fournissant ainsi des ressources précieuses pour de futures études. Un certain nombre d'ARNnc identifiés pourraient jouer un rôle dans l'adaptation de C. difficile chez l'hôte et dans la réponse immunitaire de l'hôte. Les marqueurs de l'inflammation et les cibles thérapeutiques potentielles qui ressortent de ce travail ouvrent de nouvelles voies pour les stratégies basées sur l'ARN et la modulation du microbiote afin d'améliorer l'efficacité des traitements de l'infection associée à C. difficile. -> Contact : olga.soutourina@i2bc.paris-saclay.fr

|
Scooped by
Life Sciences UPSaclay
December 28, 2024 4:47 PM
|
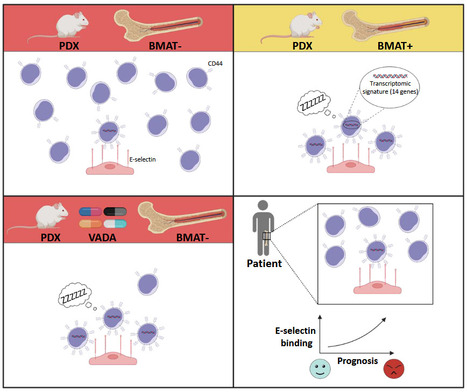
Les leucémies aiguës lymphoblastiques (LAL) sont des cancers du sang au pronostic variable selon l’âge : la survie à 5 ans dépasse 85% chez les enfants mais n’atteint que 50% chez les adultes. Cette différence pourrait s’expliquer par des facteurs physiologiques influençant la résistance des cellules leucémiques aux traitements. La moelle osseuse, niche importante des cellules leucémiques, évolue avec l’âge, l’obésité et les traitements thérapeutiques, devenant de plus en plus riche en cellules graisseuses (appelées adipocytes). Dans une étude publiée dans Leukemia, les scientifiques du Laboratoire des cellules Souches Hématopoïétiques et des Leucémies - LSHL (IRCM/CEA-Jacob/INSERM/UPSaclay, Fontenay-aux-Roses) ont identifié une population de cellules leucémiques « dormantes » exprimant fortement la protéine de surface CD44. Ces cellules sont enrichies dans la moelle osseuse riche en adipocytes de modèles précliniques et sont détectées à différents taux chez les patients, les plus élevés étant associés à un mauvais pronostic. Particulièrement insensible à la chimiothérapie, ces cellules sont dotées d’une nouvelle capacité, celle de lier une protéine appelée E-sélectine, normalement exprimée à la surface des cellules endothéliales qui tapissent les vaisseaux sanguins. Ces travaux, réalisés sur une vaste cohorte de patients, définissent l’existence de biomarqueurs (expression intense de CD44 et liaison à la E-sélectine) de la chimiorésistance des LAL. L’étape suivante sera de déterminer si cette liaison CD44/E-sélectine peut être exploitée pour cibler les cellules leucémiques échappant à la chimiothérapie, ce qui ouvrirait une nouvelle piste thérapeutique pour ces cancers du sang de pronostic sombre en cas de rechute. Lire également l'Actu CEA-Jacob -> Contact : julien.calvo@cea.fr

|
Scooped by
Life Sciences UPSaclay
December 28, 2024 5:06 PM
|
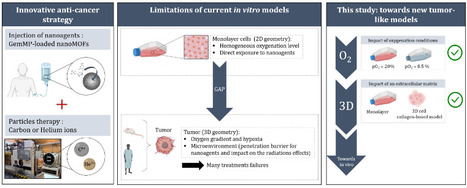
Dans une étude parue dans International Journal of Pharmaceutics, des chercheurs de l’Institut des Sciences Moléculaires d’Orsay - ISMO (CNRS/UPSaclay, Orsay) ont publié la première étude d'une stratégie innovante qui associe des nanoparticules organo-métalliques poreuses (nanoMOF) chargées du médicament anticancéreux Gemcitabine monophosphate (GemMP) et la hadronthérapie par ions hélium et carbone. Cette étude explore les effets de l'hypoxie sur la délivrance de médicaments en combinaison avec les rayonnements ionisants, démontrant que les nanoMOFs chargées de GemMP améliorent de manière significative l'efficacité anticancéreuse de la thérapie par particules dans des conditions normoxiques (pO2=20%) et hypoxiques (pO2=0,5%). Notamment, la présence de nanoMOFs chargés de GemMP permet de réduire la dose d'irradiation nécessaire à tuer 90% des cellules tumorales de 1,4 fois en normoxie et d'au moins 1,6 fois en hypoxie. D'autre part, la capacité des nanoMOFs à diffuser dans la matrice extracellulaire de modèles cellulaires en 3D et à s'accumuler dans les cellules a été mise en évidence. Un effet plus important de la GemMP encapsulée que du médicament libre a été observé, confirmant le rôle clé des nanoMOFs dans le transport de la substance active vers les cellules cancéreuses comme un cheval de Troie. Cette étide ouvre la voie à la conception de nanomédicaments « tout-en-un » où chaque composant joue un rôle dans l'optimisation du traitement du cancer afin de maximiser les effets cytotoxiques sur les cellules tumorales hypoxiques tout en minimisant la toxicité sur les tissus sains. -> Contact : erika.porcel@universite-paris-saclay.fr

|
Scooped by
Life Sciences UPSaclay
December 18, 2024 4:42 PM
|
Portrait Jeune Chercheur – Arthur T. Molines, Maître de Conférences en Biologie Cellulaire
Arthur T. Molines is a cell biologist and biophysicist who joined Paris-Saclay University as an Associate Professor in September 2024. He is also a member of the Institute for Integrative Biology of the Cell (I2BC, CNRS/CEA/UPSaclay, Gif-sur-Yvette), where he joined Christophe Le Clainche’s research team (link). His work primarily focuses on understanding the physical properties of the cytoplasm and their impact on biological functions, with a particular emphasis on the organization and dynamics of the cytoskeleton. Arthur completed his Ph.D. at Paris-Saclay University (2013–2016), where he investigated microtubule organization and dynamics in Arabidopsis thaliana. Using super-resolution and quantitative microscopy, he developed image analysis workflows to quantify the organization and dynamics of the microtubule network. His research revealed important links between microtubule organization, plant cell wall architecture, cell mechano-sensitivity, and overall plant development. He also employed in vitro reconstitution assays to compare the functions of microtubule dynamics regulators from plants and mice. After obtaining his Ph.D., Arthur joined the Department of Cell and Tissue Biology at the University of California, San Francisco (UCSF), as an independent postdoctoral scholar in Professor Fred Chang's lab (link). During this time, he became deeply interested in the cytoplasm and its physical and material properties. By applying physical perturbations and biophysical quantification techniques, he demonstrated that cytoplasm viscosity regulates cytoskeleton dynamics across a wide range of eukaryotic organisms, including yeast, plants, and mammalian cells. His research also highlighted the cytoplasm's complexity, revealing that its physical properties are heterogeneous and vary across space and between cells. Additionally, Arthur used in vitro reconstitution assays to explore how buffer composition influences microtubule dynamics. At the I2BC, Arthur continues his exploration of the physics of the cytoplasm. Using a combination of quantitative microscopy and biophysical approaches his research seeks to understand how cytoplasmic properties, such as viscosity and crowding, are regulated and how they contribute to essential cellular functions, including cell migration. As an Associate Professor at Paris-Saclay University, Arthur teaches Cell Biology and Biochemistry at both the undergraduate and master's levels. He is passionate about communicating scientific knowledge and the scientific method to students, and he enjoys sharing his enthusiasm for the sciences with them. “The first principle is that you must not fool yourself and you are the easiest person to fool.” - Richard Feynman -> Contact: arthur.molines@i2bc.paris-saclay.fr

|
Scooped by
Life Sciences UPSaclay
January 4, 11:39 AM
|
Neurologue à l’Hôpital de la Salpêtrière, Directeur de recherche à l’Inserm de l’équipe « Exposome et Hérédité » du Centre de Recherche en Epidémiologie et Santé des Populations – CESP (UMR-S 1018 INSERM/UVSQ/UPSaclay, Villejuif), Alexis Elbaz travaille sur une cohorte de près 100 000 femmes affiliées à la MGEN suivies régulièrement depuis 1990, avec l’inclusion de leurs enfants, des pères et bientôt des petits-enfants. Il étudie tout particulièrement l’épidémiologie de la maladie de Parkinson et ses interactions entre facteurs environnementaux ou comportementaux et génétiques.

|
Scooped by
Life Sciences UPSaclay
December 28, 2024 5:23 PM
|
Véronique Leblais, Chevalier dans l'ordre des Palmes Académiques
Véronique Leblais, Professeure de Pharmacologie à la Faculté de Pharmacie de l'UPSaclay, Responsable du Master DEIM et de la Mention de Master "Sciences du Médicament et des produits de santé" (UPSaclay), et co-responsable de l'équipe "Signalisation des nucléotides cycliques" au sein de l'UMR-S 1180 (Inserm/UPSaclay, Orsay), a été décorée des Palmes Académiques au titre de Chevalier le 10 décembre 2024.
|





 Your new post is loading...
Your new post is loading...