Your new post is loading...
FOCUS PLATEFORME : Appel d’offre interdisciplinaire pour un accès aux plateformes de biologie structurale, d'imagerie biologique et de protéomique
Les trois infrastructures nationales ProFi, FranceBioImaging et FRISBI ainsi que le GIS IBiSA ont le plaisir d'annoncer un 3e appel pour un accès financé aux installations labellisées IBiSA. Notre objectif est de promouvoir la mise en réseau des installations IBiSA par le biais de projets de recherche interdisciplinaires. Les candidatures doivent demander l'accès à au moins deux installations IBiSA différentes dans deux disciplines (biologie structurale, imagerie biologique et protéomique). L'appel est ouvert à tout laboratoire académique. Le projet doit être considéré comme le point de départ d'un projet plus vaste ou comme une étude de validation de concept. Le montant de l'aide financière sera de 5000 € maximum par demande pour couvrir les frais d'installation ; les frais de voyage et d'hébergement ne sont pas éligibles. Les candidatures doivent être soumises à Call-IBISA-FBI-FRISBI-PROFI@i2bc.paris-saclay.fr en utilisant le modèle de document à télécharger ICI. La date limite pour cet appel d'accès inter-infrastructures est le 31 mai 2024. Le texte complet de l’AO peut être téléchargé ICI.
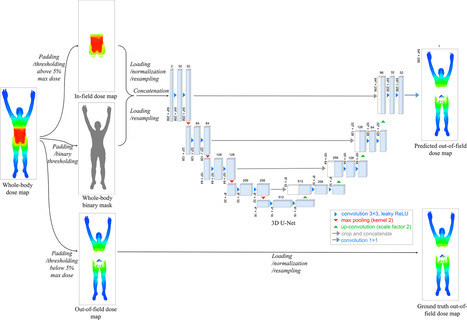
Les scientifiques de l’unité « Radiothérapie Moléculaire et Innovations Thérapeutiques » (UMR-S 1030 INSERM/UPSaclay/Gustave Roussy, Villejuif) ont publié récemment dans International Journal of Radiation Oncology Biology Physics une preuve de concept démontrant la pertinence de l’utilisation de l’intelligence artificielle pour l’estimation des doses aux corps entiers des patients lors des traitements de radiothérapie externe dans un cadre clinique. La radiothérapie externe est un traitement de lutte contre le cancer qui consiste en l’émission de particules, délivrées de façon contrôlée, permettant la destruction locale des cellules tumorales. Lors de ces traitements, des particules sont inévitablement diffusées en dehors de la zone tumorale à traiter, induisant des effets néfastes encore mal compris, comme l’apparition de seconds cancers ou des dysfonctionnements immunologiques. Par manque d’outils compatibles, ces doses déposées à distance de la zone à traiter ne sont pas estimées en routine clinique. Avec cette preuve de concept, les auteurs démontrent pour la première fois que l’intelligence artificielle, et plus spécifiquement qu’un réseau de neurones est un outil pertinent pour l’estimation rapide, précise et polyvalente de la dose à distance, compatible avec la prise en charge clinique actuelle. Un U-Net 3D a été entraîné sur plus de 3000 cartes de doses corps entiers de patients variées. Des performances en très bonne cohérence avec la littérature actuelle sont démontrées, associées à des temps d’inférence réduits, ce qui est inédit pour ce genre d’application. Cet outil ouvre la voie à des études à plus grande échelle des effets des doses à distance, et permet également d'envisager la considération de ces faibles doses lors de la prise en charge des patients. Contact : nathan.benzazon@gustaveroussy.fr
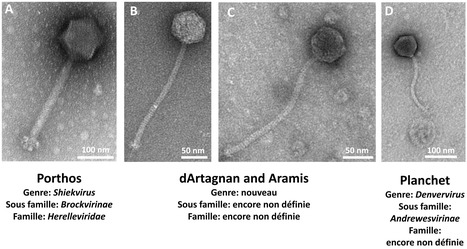
Les bactériophages (phages) virulents, virus bactériens qui infectent et lysent obligatoirement leurs hôtes, représentent une alternative sérieuse pour lutter contre les infections bactériennes multirésistantes aux antibiotiques. Cependant, le spectre d’hôtes d’un bactériophage, c’est-à-dire la gamme de bactéries qu’il est capable d’infecter, est souvent restreint à quelques souches d’une même espèce. Une telle spécificité d’action peut être un inconvénient de la phagothérapie car elle nécessite de disposer d’un phage efficace contre la souche bactérienne responsable de l’infection. Étant donné la capacité évolutive des phages, il est envisageable d’anticiper cette difficulté, en faisant évoluer in vitro leurs spectres d’hôtes vers un plus large panel de souches cliniques. La faisabilité d’une telle approche sur des isolats cliniques d’Enterococcus faecium, vient d’être publiée dans Antimicrobial Agents and Chemotherapy, par des scientifiques de l’Institut Micalis (INRAE/AgroParisTech/UPSaclay, Jouy-en-Josas), en collaboration avec leurs collègues du CHU de Rennes. E. faecium est l’un des pathogènes opportunistes dont les niveaux de résistance aux antibiotiques sont parmi les plus préoccupants à l’échelle mondiale. L’évolution dirigée de quatre bactériophages, essentiellement isolés d’eaux usées, combinés en cocktail sur des souches cliniques de l’espèce a permis d’isoler plusieurs phages dérivés ayant des spectres d’hôtes élargis. Le meilleur phage évolué est capable de s’attaquer à 10 des 14 souches cliniques testées, contre 5 pour son ancêtre non évolué. Son spectre d’action couvre ainsi le double de clones hyper-épidémiques, sans qu’il se soit élargi aux espèces proches, limitant les risques d’impact collatéral. Le génome des phages évolués contient toujours diverses mutations ponctuelles ou des remaniements dans la région génétique des fibres caudales, connues pour jouer un rôle clef dans la reconnaissance de l’hôte bactérien par les phages. En conclusion, une démarche anticipatrice, consistant à isoler des phages de l’environnement, puis à les « entraîner » sur des collections de souches cliniques problématiques devrait, à terme, pouvoir faciliter l’usage raisonné des phages en cas d’impasse thérapeutique. Légende Figure : Quatre bactériophages sélectionnés pour un entrainement en vue d’accroître leurs spectres d’hôtes sur des souches cliniques d’E. faecium. Ils sont ici examinés en microscopie électronique en transmission et correspondent à des phages caudés appartenant à la classe des Caudoviricetes. Ils représentent trois genres, sous familles et familles phagiques différentes. Pour connaître précisément le programme d’entrainement, lequel de ces phages y a le mieux répondu et pourquoi le quatrième mousquetaire, le phage Athos, n’y a pas participé. Contact : julien.lossouarn@inrae.fr
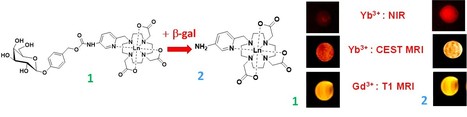
Dans une étude publiée dans Angewandte Chemie International Edition, des scientifiques du département de chémobiologie de l’Institut de chimie des substances naturelles - ICSN (CNRS/UPSaclay, Gif-sur-Yvette), en collaboration avec deux équipes du Centre de Biophysique Moléculaire (CBM) d’Orléans, ont conçu des complexes de lanthanides (Ln3+), dont la structure peut être modifiée par l’activité catalytique d’une enzyme. Cette modification induit une variation de leur signal de luminescence proche infrarouge (NIR), ainsi que de celui observé en imagerie de résonance magnétique (IRM) : IRM-CEST (Transfert de Saturation par échanges chimiques) et en IRM-T1 classique, en fonction de la nature du Ln3+ utilisé. Les auteurs ont ainsi démontré qu’il était possible de suivre l'activité de la β-galactosidase en fonction du temps par l’imagerie de luminescence proche-infrarouge et par IRM-CEST dans des fantômes contenant le complexe d’ytterbium (Yb3+) et en IRM-T1 avec le complexe de gadolinium (Gd3+). Ces résultats ouvrent la voie vers le suivi de biomarqueurs enzymatiques au moyen de plusieurs modalités d’imagerie complémentaires en utilisant une sonde de structure moléculaire unique, évitant les biais associés à l’utilisation de plusieurs sondes. La conception modulaire de ces complexes permet d’envisager dans le futur la détection d’autres cibles enzymatiques d’intérêt, par simple modification de l’entité sensible à l’enzyme tout en conservant le même module de détection. Contact : philippe.durand@cnrs.fr
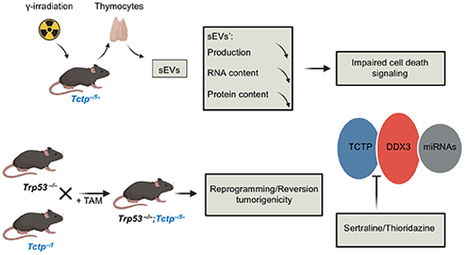
L’équipe d’Adam Telerman et Robert Amson dans l’UMR-S 981 (INSERM/UPSaclay/Gustave Roussy, Villejuif) travaille depuis plus de trente ans sur la réversion/reprogrammation tumorale. Leurs modèles biologiques ont permis d’identifier TCTP (Translationally Controlled Tumor Protein) comme l’un des gènes régulateurs du programme de réversion tumorale. Dans un article publié dans EMBO reports, l’équipe a étudié le rôle de TCTP dans la communication intercellulaire. En utilisant des souris Tctp knockout conditionnelles qu’ils ont générées, Ils démontrent que les thymocytes sont déficients dans leurs réponses à l’irradiation dans le cadre du stress génotoxique. L’analyse des nano-vésicules (small extracellular vesicles: sEVs) de ces thymocytes délétés dans Tctp indique une diminution importante de leur sécrétion. De plus, leur contenu en protéines et ARN est aussi diminué. Ces sEVs ne communiquent plus l’apoptose aux cellules environnantes (Bystander effect). D’autre part, la génération de ces souris déficientes en Tctp a également permis d’investiguer le rôle joué par ce gène dans le cancer. Il est bien connu que des souris mutées dans le gène p53 meurent de cancer dans les 29 semaines. En revanche, ces mêmes souris p53 mutantes délétées dans TCTP ont une survie significativement augmentée, à plus de 60 semaines. Ces résultats permettent pour la première fois d’investiguer le rôle de TCTP et des sEVs dans un modèle in vivo tant sur le transfert du signal apoptotique que dans la tumorigénicité. Contact : atelerman@gmail.com ou amson.robert@gmail.com
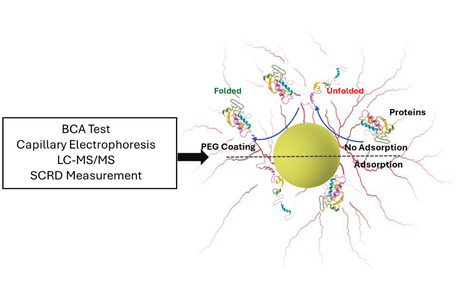
Les nanomédicaments, capables de protéger et transporter des molécules actives jusqu’à leur cible biologique, permettent des avancées majeures dans le traitement de maladies, comme les infections sévères ou le cancer. Cependant, une compréhension incomplète de l’interaction entre les nanomédicaments et le milieu vivant, en particulier les protéines plasmatiques, constitue un obstacle majeur à leur développement et transposition en clinique. En effet, les interactions nano-bio déterminent le devenir, l'efficacité et l'immunotoxicité in vivo des nanomédicaments. Ces interactions peuvent également altérer l’activité biologique des protéines plasmatiques. Pour répondre au besoin croissant d'étudier les interactions nano-bio, les scientifiques de l’ISMO et de l’IGPS (CNRS/UPSaclay, Orsay) ont établi une approche orthogonale afin de déterminer : (i) la formation de couronnes protéiques et (ii) les modifications induites par les nanomédicaments sur la structure et la stabilité des protéines. Dans leur étude publiée dans Drug Delivery and Translationnal Research, ils utilisent cette méthodologie pour analyser ces interactions, qualitativement et quantitativement, en utilisant l'albumine du sérum humain (HSA) comme protéine modèle. La chromatographie liquide-spectrométrie de masse et l’électrophorèse capillaire de zone (CZE) ont permis de déterminer la quantité et la stabilité des protéines adsorbées. La CZE a permis d’obtenir des informations qualitatives sur les différentes formes de HSA (native, glyquée et cystéinylée). La structure secondaire et la stabilité thermique de la HSA ont été évaluées à l’aide du dichroïsme circulaire utilisant le rayonnement synchrotron. Des études comparatives des nanoparticules de divers types (organiques ou hybrides) recouvertes ou non de polyéthylène glycol (PEG) ont montré l’effet protecteur des chaines de PEG. Les auteurs ont également démontré que même sans adsorption de la HSA à la surface des nanoparticules, son interaction avec les nanomédicaments peut provoquer une modification de sa structure. Cette étude fait partie d’une collaboration de longue date entre les équipes complémentaires du Dr. Ruxandra Gref (ISMO) et du Pr. Claire Smadja (IGPS). Contact : ruxandra.gref@universite-paris-saclay.fr ou claire.smadja@universite-paris-saclay.fr
Les petits-déjeuners de l’écosystème PSCC : Rencontrez les acteurs de l’innovation en oncologie ! Découvrez leur expertise et leur offre aux projets innovants Nous avons le plaisir de vous inviter à participer au « petit-déjeuner de l’écosystème » du PSCC, qui aura lieu mardi 30 avril 2024 en mode hybride. A l’occasion de cet événement, nous serons ravis d’accueillir Lionel Riou-Franca, RWE Chief Scientific Officer chez AIXIAL Group qui interviendra sur le thème « RWD / RWE (Real-World Data / Real-World Evidence) ». Ne manquez pas cette rencontre dans le cadre d’un petit-déjeuner convivial propice aux échanges. Pour vous inscrire, merci de suivre ce lien
Le prochain Meeting de l’EUR Saclay Plant Sciences (SPS) aura lieu les 3 et 4 juin 2024 sur le Campus Agro Paris-Saclay (Palaiseau). Le 4 juin sera une journée scientifique ouverte à tous. L’inscription à cette journée scientifique est gratuite mais obligatoire La date limite pour s’inscrire est le 16 mai 2024 (minuit). Programme prévisionnel du 4 juin : 09:00 AM Welcome coffee
09:20 AM Introduction (Loïc Lepiniec)
09:30 AM Title to come - Claudia Jonak (Austrian Institute of Technology, Austria)
10:15 AM Title to come (SAB member)
11:00 AM Coffee break
11:30 AM The study of wild and domestic forms provides a dual perspective on the evolution of divergence with gene flow - Maud Tenaillon (UMR Quantitative Genetics and Evolution - GQE-Le Moulon) 12:15 PM Lunch (offered by SPS) 01:45 PM Unveiling the functional mechanism of Fatty Acid Photodecarboxylase: an algal photoenzyme with great promise for green fuel production - Pavel Müller (Institute of Integrative Biology of the Cell - I2BC)
02:30 PM The colorful genetic makeup of Whiteflies sheds light on plant-insect interactions - Florian Maumus (Unité de Recherche en Génomique-Info - URGI)
03:15 PM How plants deal with replication-induced DNA damage: signaling mechanisms and physiological relevance - Cécile Raynaud (Institut of Plante Sciences Paris-Saclay - IPS2)
04:00 PM Coffee break
04:30 PM Into outer space: fungal secretion of extracellular vesicles and their role in plant infection - Richard O'Connell (UMR BIOlogie et GEstion des Risques en agriculture - BIOGER)
05:15 PM Spatio-temporal regulation of plant cell division - David Bouchez (Institut Jean-Pierre Bourgin - IJPB) 06:00 PM End of the meeting
Claude Desplan - Institutional seminar "Mayent - Rothschild", 2 mai 2024 à 11h30 (Orsay)
Mayent-Rothschild seminar Claude Desplan Thursday, May 2nd 2024, 11:30 am Centre de recherche - Orsay Amphithéâtre du Bâtiment 111 Claude Desplan is Silver Professor at NYU and a specialist of neural diversity. He has produced very elegant work with his lab over the last 20 years trying to understand the mechanisms of stochastic choices during developmental processes. He then moved to unravel the process of neural diversity during the formation of the optic medulla using Drosophila as a model. He more recently studied the fascinating process of aging and rejuvenation in social insects (see his lab web page). Claude Desplan received many awards for his remarkable work, including the Conklin medal, and is an elected member of the National Academy of Science, USA. In the Drosophila optic lobes, ~250 neuronal types organized as 800 columns process the inputs from 800 unit-eyes. Neural stem cells in the medulla sequentially express a series of temporal transcription factors (tTFs), producing at each temporal window different neurons that innervate each of the 800 columns. We used single-cell mRNA sequencing to identify the tTFs that specify most medulla neurons. Each tTF regulates the progression of the series by activating the next tTF and repressing the previous one. Furthermore, the neuroepithelium that generates these stem cells is patterned into subdomains by spatial TFs: Although the series of tTFs is the same in stem cells originating from all spatial domains, the neurons they produce differ. Therefore, the integration of temporal and spatial patterning as well as Notch status, are sufficient to explain the generation of the entire neuronal diversity in this brain region. Finally, we will show how diversity in the brain can evolve to affect specific sensory functions in different species. I will describe the dramatically increased diversity in the mushroom body in ants as compared to flies, as ants rely extensively on pheromones rather than vision for their eusocial life. Contact(s): Carsten.Janke@curie.fr ou pierre.leopold@curie.fr
Les ministères des Affaires étrangères et de la Recherche soutiennent des coopérations scientifiques entre la France et le Chili.
L'Inserm finance à hauteur de 10 000 € des collaborations entre des chercheurs ou des chercheuses Inserm (CRCN depuis moins de 5 ans, ATIP-Avenir ou chaires Inserm) et une équipe étrangère.
La Graduate School Health & Drug Sciences lance un appel à projet "Junior Project Leader". Il s'adresse aux chercheurs, enseignants-chercheurs et ingénieurs de recherche titularisés depuis moins de 10 ans. Il a pour objectif d'encourager l'émergence de thématiques propres aux lauréats et de favoriser leur autonomisation.
Le programme "recherche à risque" EXPLOR'AE, confié à INRAE par le Ministère de la Recherche et de l'enseignement supérieur, est financé par France 2030 à hauteur de 20 M€ pour la première phase. Il vise à soutenir des recherches associant risque, rupture et impact, à accompagner les talents et les idées, à créer les conditions pour générer des ruptures conceptuelles ou technologique et ainsi contribuer à répondre aux grands défis sociétaux et aux transitions associées (agroécologique, énergétique, numérique et de santé). Il est destiné à l'ensemble de la communauté scientifique nationale du périmètre agriculture, alimentation et environnement. Voir les actions en cours
|
Des collègues scientifiques et non-scientifiques de l’Institut Pasteur, du CEA et de l’Université Paris-Saclay organisent un évènement public les vendredi 24 et samedi 25 mai 2024 à l’Institut Pasteur (gratuit, sur inscription). Cet évènement - ReCombinaisons, science, fiction, société - fera se rencontrer des chercheuses et chercheurs en biologie-santé, en science humaines et sociales et en philosophie, des auteurs et autrices de science-fiction, des personnes qualifiées, et du public pour discuter de thèmes de recherche et de société, avec un accent sur la place et les évolutions de la recherche dans la société, dans un contexte qui devient de plus en plus complexe. ReCombinaisons est organisé autour d’une dizaine de tables-rondes pendant lesquelles la modératrice ou le modérateur fera discuter entre eux les intervenantes et intervenants pendant 45 minutes, puis avec le public pendant 15 à 20 minutes supplémentaires. Il y aura aussi deux conférences, la projection d’un documentaire, et un atelier participatif sur la conception de jeux vidéo scientifiques. Nous avons cherché à favoriser la diversité des approches et des regards en privilégiant des tables inter-disciplinaires avec un apport de la science-fiction et en essayent de réunir des gens pouvant porter des regards décentrés sur les questions abordées.
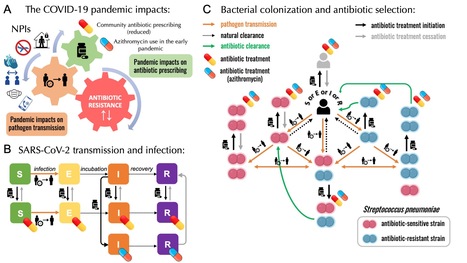
Au début de l'année 2020, le monde a été témoin d'interventions non pharmaceutiques sans précédent pour lutter contre la transmission du SARS-CoV-2. Il est intéressant de noter que ces mesures ont également eu des effets inattendus sur l'incidence des maladies pneumococciques invasives. Alors que des réductions globales des maladies invasives telles que la bactériémie et la méningite ont été observées, la plupart des pays européens ont signalé une augmentation préoccupante de la résistance aux antibiotiques parmi les isolats de Streptococcus pneumoniae invasifs entre 2019 et 2020. Pour compliquer la situation, des études ont révélé que les taux de portage de pneumocoques sont restés largement stables au cours de cette même période. Dans une étude publiée dans eLife, des scientifiques du Laboratoire Epidémiologie et modélisation de la résistance aux antimicrobiens (Centre de Recherche en Epidémiologie et Santé des Populations – CESP Institut Pasteur/UParis Cité/UPSaclay/UVSQ/Inserm), en collaboration avec des scientifiques du Conservatoire national des arts et métiers (MESuRS), ont mis au point un modèle mathématique de transmission simultanée du SARS-CoV-2 et des souches de S. pneumoniae sensibles et résistantes aux antibiotiques, qui permet d'explorer cette interaction complexe. Il est surprenant de constater que seuls deux des 31 scénarios de pandémie testés ont pu reproduire les tendances observées dans la population générale. Ces scénarios ont identifié la réduction de l'utilisation globale d'antibiotiques, l'absence de virus communs et le traitement à l'azithromycine dans les cas de COVID-19 comme des mécanismes clés ayant un impact sur les tendances pneumococciques pendant la pandémie. Contact : aleksandra.kovacevic@pasteur.fr
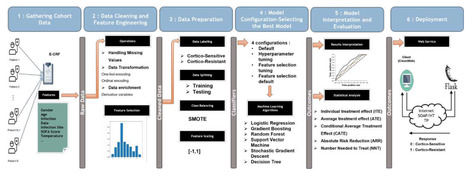
Dans une étude publiée dans Computer Methods and Programs in Biomedicine, les chercheurs du laboratoire d’informatique DAVID (UVSQ/UPSaclay, Versailles) ont examiné la sensibilité aux corticostéroïdes chez les patients septicémiques. Le sepsis, une infection grave potentiellement mortelle, peut bénéficier du traitement par corticostéroïdes, mais certains patients présentent une résistance à ce traitement. Pour anticiper cette réponse, les chercheurs ont développé une méthodologie de fouille de données. En utilisant les données de l'essai contrôlé randomisé APROCCHSS, comprenant 1241 patients septicémiques (Service de Réanimation médico-chirurgicale, hôpital Raymond-Poincaré, AP-HP, Garches), ils ont créé un modèle de prédiction efficace, appelé la "signature", basé sur la régression logistique. Ce modèle a démontré une AUC de 72%, et des expérimentations supplémentaires ont examiné l'impact des caractéristiques supplémentaires et la généralisabilité du modèle à différents groupes de patients. Des analyses statistiques ont également évalué l'effet du traitement à la fois au niveau individuel et populationnel. Cette méthodologie a été intégrée dans le système d'information de l'Assistance Publique - Hôpitaux de Paris (AP-HP) sous la forme d'un service web, permettant aux cliniciens de prédire la sensibilité ou la résistance aux corticostéroïdes chez les patients septicémiques et d'intervenir précocement pour améliorer les résultats cliniques. Contact : rahma.hellali@uvsq.fr
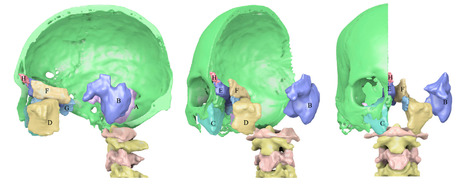
Dans une étude publiée dans Clinical Anatomy, les scientifiques du Laboratoire Anthropologie, Archéologie, Biologie - LAAB (UFR Simone Veil - santé UVSQ/UPSaclay) ont étudié les sinus de la face de la tête embaumée du roi de France Henri IV. La tête conservée du roi Henri IV de France (1553-1610) a survécu jusqu'à nos jours grâce à un embaumement de haute qualité et à des conditions de conservation favorables. Le but de cette étude était d’examiner les cavités résonantes supérieures et les cellules mastoïdiennes d’Henri IV à l’aide d’une méthode médicale de segmentation 3D originale et innovante mise au point par le LAAB en lien avec le Laboratoire de Phonétique et Phonologie de l’Université Sorbonne Nouvelle/Paris 3 (UMR 7018). Les sinus paranasaux et les cellules mastoïdiennes du roi Henri IV ont été étudiés en croisant les informations biographiques disponibles avec un examen endoscopique flexible (à l'hôpital Foch) et une imagerie par tomodensitométrie (TDM). Les sinus paranasaux et les cellules mastoïdiennes ont été délimités et leurs volumes ont été évalués à l'aide du logiciel ITK-SNAP 4.0 (open source). La tomodensitométrie des sinus paranasaux a révélé des anomalies de forme et de nombre. Henri IV de France souffrait d'aplasie des sinus. Ni le sphénoïde gauche ni le sinus frontal gauche ne contrastaient nettement et une pneumatisation remarquable des processus clinoïdes droits s'étendait sur toute la hauteur du processus ptérygoïdien droit. Les volumes totaux des alvéoles mastoïdiennes d'Henri IV ont été estimés respectivement à 27 et 26 mL pour les côtés droit et gauche, dépassant largement la moyenne normale et le maximum des sujets modernes. Aucun signe d’affection chronique des oreilles ou des sinus n’a été mis en évidence. Au total, en se fondant sur ce cas bio-historique, une nouvelle méthode a été développée pour établir des hypothèses sur la croissance et les conditions respiratoires du visage. C'est un prérequis nécessaire pour la reconstitution de la voix de ce personnage historique, un travail en cours au sein du LAAB. Contact : philippe.charlier@uvsq.fr
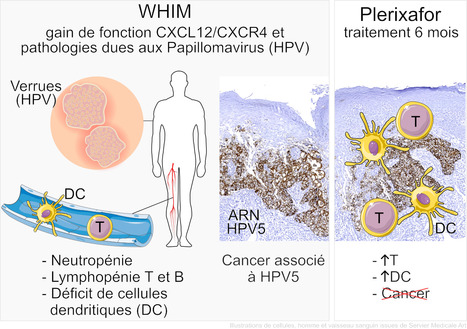
Dans une étude publiée dans New England Journal of Medicine, les scientifiques de l’équipe « Immunoregulation, Chemokines and Viral Persistence » de l’UMR-S 996 (INSERM/UPSaclay, Orsay) et leurs collaborateurs des hôpitaux de Nancy, du Kremlin-Bicêtre et du centre de Référence des Neutropénies Chroniques, rapportent les effets d’un traitement expérimental basé sur un antagoniste du récepteur CXCR4 de la chimiokine CXCL12 ; le Plerixafor fourni par Sanofi–Genzyme. Le patient concerné est atteint d’un syndrome d’immunodéficience très rare (WHIM) causé par un gain-de-fonction de CXCL12/CXCR4 qui se manifeste par une profonde leucopénie et une susceptibilité sélective au développement de pathologies dues aux papillomavirus (HPV), sans traitement spécifique à ce jour. Ces virus (≈440) strictement épithéliaux font partie du microbiote cutané commensal chez chacun d’entre nous et se répliquent généralement de manière asymptomatique. Ils sont cependant dotés d’un potentiel pathogène qui peut se manifester par des dysplasies légères (verrues). Non contrôlées, ces dysplasies peuvent évoluer dans de rares cas vers des cancers. Le traitement a entrainé l’augmentation des leucocytes et la disparition du carcinome anal, associé de manière inattendue à un HPV cutané. Ces résultats confirment l’importance de l’axe CXCL12/CXCR4 dans le contrôle des infections HPV, constituent un argument important en faveur du traitement chronique ciblant CXCR4 chez les patients WHIM et, plus généralement, les immunodéprimés sensibles aux pathologies HPV, et alertent sur l’importance de tests de typage des HPV à large spectre pour les carcinomes anogénitaux. Contact : viviana.marin-esteban@universite-paris-saclay.fr ou francoise.bachelerie@universite-paris-saclay.fr ou claire.deback@universite-paris-saclay.fr
La Fédération d’Hématologie de l’Université Paris-Saclay, créée en 2022, regroupe les équipes d'hématologie expérimentale du CEA de Fontenay aux Roses, de Gustave Roussy à Villejuif, des hôpitaux de Bicêtre et Paul Brousse et de l'institut Curie à Orsay. Elle organise pour la première fois une école d’été du 1er au 3 juillet 2024. Des cours théoriques portant sur l’hématopoïèse normale et pathologique seront dispensés par des chercheurs/médecins/biologistes affiliés à l’UPS durant 2 jours. La 3ème journée sera consacrée à des travaux pratiques qui seront réalisés dans les laboratoires de Gustave Roussy et du CEA de Fontenay aux roses. Par cette école d’été, nous souhaitons offrir à des étudiants de Licence 3 ou Master 1 de l’UPSaclay la possibilité de découvrir/approfondir leurs connaissances en hématologie. Il s’agit d’une opportunité unique d’échanger avec des experts internationalement reconnus en recherche fondamentale ou translationnelle à travers un format qui facilitera les échanges. Grâce à cette formation, les étudiants pourront mieux identifier les thématiques ainsi que les interlocuteurs utiles pour la poursuite de leurs études dans le domaine de l’hématologie au sein de l’UPSaclay. Pour candidater, veuillez compléter le formulaire en ligne avant le 3 mai 2024. Si vous souhaitez plus de détails, n’hésitez pas à scanner le QR code sur le flyer de présentation ci-dessus, à vous rendre sur le site de la fédération ou à nous contacter à l’adresse mail suivante : emilie.elvira-matelot@inserm.fr Le Comité d’organisation
La Graduate School Métiers de la Recherche et de l’Enseignement Supérieur (GS MRES) vous a déjà annoncé sa troisième édition du congrès junior pluridisciplinaire destiné aux étudiantes et aux étudiants de l’ensemble de l’Université Paris-Saclay (composantes, établissements-composantes et universités associées) et de toutes les disciplines. Le but est de venir découvrir la diversité des disciplines et des formations rassemblées dans l’Université Paris-Saclay à travers la présentation des travaux de recherche auxquels les étudiantes et les étudiants prennent part au sein des formations. Congrès Junior Pluridisciplinaire jeudi 23 mai 2024 après-midi (et soir) à l’ENS Paris-Saclay Tout comme les 2 années précédentes, les étudiantes et les étudiants ont été nombreux à demander une participation au congrès. Le comité scientifique, composé de membres des différentes Graduate Schools, travaille actuellement à l’étude des demandes et va sélectionner les présentations orales, les posters et les vidéos pré-enregistrées qui seront visibles le jour du congrès. Il devrait y avoir environ 50 présentations orales de 5 minutes, 10 vidéos pré-enregistrées de 5 minutes et 30 posters. L’ensemble des composantes, établissements-composantes et universités associées de l’Université Paris-Saclay, l’ensemble des Graduate Schools, ainsi qu’un grand nombre de disciplines seront fièrement représentées le jour du congrès. Le déroulé de la demi-journée et le programme avec tous les intervenants seront bientôt présents sur le site du congrès. Pour venir assister aux présentations, il faut compléter le « formulaire d’inscription en tant que spectateur / spectatrice » accessible sur le site du congrès (ne pas compléter le « formulaire de demande de participation ») : Les inscriptions fermeront le 26 avril 2024, ne tardez pas ! Que vous soyez étudiant participant ou non, chercheur, enseignant, enseignant-chercheur, administratif, famille ou autre, nous espérons avoir le plaisir de vous compter parmi nous le 23 mai 2024 pour soutenir les étudiantes et étudiants qui présentent et pour découvrir l’ensemble de leurs travaux. Le comité d’organisation du congrès junior de la GS MRES
SAVE THE DATE ! RIC Réseau d'Imagerie Cellulaire Paris-Saclay - Demi-journée Super résolution - 3 octobre 2024
Demi-journée consacrée à l'imagerie super résolutive organisée par le Réseau d'Imagerie Cellulaire de Paris-Saclay et qui aura lieu le 3 octobre 2024 dans l'auditorium du bâtiment recherche de la Faculté de Médecine UPSaclay au Kremlin Bicêtre.
It is our great pleasure to announce the 7th edition of the « Physics and Biological Systems » biennal conference PhysBio2024 taking place on the 26-28th of June 2024 at Ecole Polytechnique, Palaiseau.
As usual, this conference aims to bring together a broad range of physical and life scientists working at the interface between the two disciplines around in-depth talks by first-rate international speakers.
The registration is open HERE at a discount price (early registration) before the 24th of May 2024, and regular price after.
The number of attendees will be limited to 200.
We hope to see you there !
The organizing committee.
Le programme Projets de recherche internationaux de l'Inserm permet de consolider des collaborations avec des partenaires étrangers. Il finance des projets de recherche à hauteur de 12 000 à 15 000 € par an sur 5 ans.
La Graduate School Health & Drug Sciences lance son appel à projet "New collaborations". Il vise à soutenir et à promouvoir la recherche en innovation thérapeutique en initiant de nouveaux projets collaboratifs entre au moins 2 équipes de recherche de la GS (hors plateforme).
Le plateau de phénotypage in vivo et transparisation organise sa 2ème journée d’animation scientifique le 12 juin 2024 à l'INRAE de Jouy-en-Josas. À cette occasion, les scientifiques utilisateurs du plateau présenteront les projets menés au sein du plateau pour illustrer leurs diversités ainsi que les expertises et techniques de phénotypage utilisées pour leurs applications. Cette journée a pour vocation de promouvoir les échanges entre utilisateurs et au-delà puisque l’événement est ouvert à tous. Nous vous présenterons également les activités de R&D conduites cette année et les projets en cours. L'inscription est gratuite mais obligatoire en suivant ce lien (il vous suffit de cliquer sur "inscription" dans le menu de gauche puis de vous connecter avec votre compte HAL ou Sciencesconf. Si vous n'en possédez pas alors il faut en créer un en cliquant sur "je n'ai pas de compte").
Symposium international IJPB 2024 "Modélisation des plantes : Opportunités et défis" organisé par de l’Institut Jean-Pierre Bourgin – IJPB à Versailles du 13 au 15 mai 2024 Le symposium IJPB 2024 comportera trois conférences plénières et quatre sessions thématiques traitant de la modélisation des plantes à différentes échelles, du niveau intracellulaire au niveau macroscopique. Le symposium comprendra également des ateliers et une présentation des infrastructures de l'IJPB, en collaboration avec la plateforme d'imagerie et de microscopie de l'Observatoire des plantes et les équipes "Modélisation et imagerie numérique" et "Paroi cellulaire primaire" de l'IJPB. Les présentations seront données par des orateurs invités et par des participants sélectionnés sur la base des résumés soumis. Ce symposium sera une occasion unique d'évaluer les avancées récentes en matière de modélisation, d'explorer la manière dont elle peut être intégrée expérimentalement et d'examiner comment elle s'adapte à l'augmentation constante de la puissance de calcul et à l'explosion des données. Les inscriptions sont ouvertes. Site internet de la manifestation
Via Life Sciences UPSaclay
|





 Your new post is loading...
Your new post is loading...