Your new post is loading...
FOCUS PLATEFORME : Appel d’offre interdisciplinaire pour un accès aux plateformes de biologie structurale, d'imagerie biologique et de protéomique
Les trois infrastructures nationales ProFi, FranceBioImaging et FRISBI ainsi que le GIS IBiSA ont le plaisir d'annoncer un 3e appel pour un accès financé aux installations labellisées IBiSA. Notre objectif est de promouvoir la mise en réseau des installations IBiSA par le biais de projets de recherche interdisciplinaires. Les candidatures doivent demander l'accès à au moins deux installations IBiSA différentes dans deux disciplines (biologie structurale, imagerie biologique et protéomique). L'appel est ouvert à tout laboratoire académique. Le projet doit être considéré comme le point de départ d'un projet plus vaste ou comme une étude de validation de concept. Le montant de l'aide financière sera de 5000 € maximum par demande pour couvrir les frais d'installation ; les frais de voyage et d'hébergement ne sont pas éligibles. Les candidatures doivent être soumises à Call-IBISA-FBI-FRISBI-PROFI@i2bc.paris-saclay.fr en utilisant le modèle de document à télécharger ICI. La date limite pour cet appel d'accès inter-infrastructures est le 31 mai 2024. Le texte complet de l’AO peut être téléchargé ICI.
Les petits-déjeuners de l’écosystème PSCC : Rencontrez les acteurs de l’innovation en oncologie ! Découvrez leur expertise et leur offre aux projets innovants Nous avons le plaisir de vous inviter à participer au « petit-déjeuner de l’écosystème » du PSCC, qui aura lieu mardi 30 avril 2024 en mode hybride. A l’occasion de cet événement, nous serons ravis d’accueillir Lionel Riou-Franca, RWE Chief Scientific Officer chez AIXIAL Group qui interviendra sur le thème « RWD / RWE (Real-World Data / Real-World Evidence) ». Ne manquez pas cette rencontre dans le cadre d’un petit-déjeuner convivial propice aux échanges. Pour vous inscrire, merci de suivre ce lien
Le prochain Meeting de l’EUR Saclay Plant Sciences (SPS) aura lieu les 3 et 4 juin 2024 sur le Campus Agro Paris-Saclay (Palaiseau). Le 4 juin sera une journée scientifique ouverte à tous. L’inscription à cette journée scientifique est gratuite mais obligatoire La date limite pour s’inscrire est le 16 mai 2024 (minuit). Programme prévisionnel du 4 juin : 09:00 AM Welcome coffee
09:20 AM Introduction (Loïc Lepiniec)
09:30 AM Title to come - Claudia Jonak (Austrian Institute of Technology, Austria)
10:15 AM Title to come (SAB member)
11:00 AM Coffee break
11:30 AM The study of wild and domestic forms provides a dual perspective on the evolution of divergence with gene flow - Maud Tenaillon (UMR Quantitative Genetics and Evolution - GQE-Le Moulon) 12:15 PM Lunch (offered by SPS) 01:45 PM Unveiling the functional mechanism of Fatty Acid Photodecarboxylase: an algal photoenzyme with great promise for green fuel production - Pavel Müller (Institute of Integrative Biology of the Cell - I2BC)
02:30 PM The colorful genetic makeup of Whiteflies sheds light on plant-insect interactions - Florian Maumus (Unité de Recherche en Génomique-Info - URGI)
03:15 PM How plants deal with replication-induced DNA damage: signaling mechanisms and physiological relevance - Cécile Raynaud (Institut of Plante Sciences Paris-Saclay - IPS2)
04:00 PM Coffee break
04:30 PM Into outer space: fungal secretion of extracellular vesicles and their role in plant infection - Richard O'Connell (UMR BIOlogie et GEstion des Risques en agriculture - BIOGER)
05:15 PM Spatio-temporal regulation of plant cell division - David Bouchez (Institut Jean-Pierre Bourgin - IJPB) 06:00 PM End of the meeting
Claude Desplan - Institutional seminar "Mayent - Rothschild", 2 mai 2024 à 11h30 (Orsay)
Mayent-Rothschild seminar Claude Desplan Thursday, May 2nd 2024, 11:30 am Centre de recherche - Orsay Amphithéâtre du Bâtiment 111 Claude Desplan is Silver Professor at NYU and a specialist of neural diversity. He has produced very elegant work with his lab over the last 20 years trying to understand the mechanisms of stochastic choices during developmental processes. He then moved to unravel the process of neural diversity during the formation of the optic medulla using Drosophila as a model. He more recently studied the fascinating process of aging and rejuvenation in social insects (see his lab web page). Claude Desplan received many awards for his remarkable work, including the Conklin medal, and is an elected member of the National Academy of Science, USA. In the Drosophila optic lobes, ~250 neuronal types organized as 800 columns process the inputs from 800 unit-eyes. Neural stem cells in the medulla sequentially express a series of temporal transcription factors (tTFs), producing at each temporal window different neurons that innervate each of the 800 columns. We used single-cell mRNA sequencing to identify the tTFs that specify most medulla neurons. Each tTF regulates the progression of the series by activating the next tTF and repressing the previous one. Furthermore, the neuroepithelium that generates these stem cells is patterned into subdomains by spatial TFs: Although the series of tTFs is the same in stem cells originating from all spatial domains, the neurons they produce differ. Therefore, the integration of temporal and spatial patterning as well as Notch status, are sufficient to explain the generation of the entire neuronal diversity in this brain region. Finally, we will show how diversity in the brain can evolve to affect specific sensory functions in different species. I will describe the dramatically increased diversity in the mushroom body in ants as compared to flies, as ants rely extensively on pheromones rather than vision for their eusocial life. Contact(s): Carsten.Janke@curie.fr ou pierre.leopold@curie.fr
Les ministères des Affaires étrangères et de la Recherche soutiennent des coopérations scientifiques entre la France et le Chili.
L'Inserm finance à hauteur de 10 000 € des collaborations entre des chercheurs ou des chercheuses Inserm (CRCN depuis moins de 5 ans, ATIP-Avenir ou chaires Inserm) et une équipe étrangère.
La Graduate School Health & Drug Sciences lance un appel à projet "Junior Project Leader". Il s'adresse aux chercheurs, enseignants-chercheurs et ingénieurs de recherche titularisés depuis moins de 10 ans. Il a pour objectif d'encourager l'émergence de thématiques propres aux lauréats et de favoriser leur autonomisation.
Le programme "recherche à risque" EXPLOR'AE, confié à INRAE par le Ministère de la Recherche et de l'enseignement supérieur, est financé par France 2030 à hauteur de 20 M€ pour la première phase. Il vise à soutenir des recherches associant risque, rupture et impact, à accompagner les talents et les idées, à créer les conditions pour générer des ruptures conceptuelles ou technologique et ainsi contribuer à répondre aux grands défis sociétaux et aux transitions associées (agroécologique, énergétique, numérique et de santé). Il est destiné à l'ensemble de la communauté scientifique nationale du périmètre agriculture, alimentation et environnement. Voir les actions en cours
Portrait Jeune Chercheuse - Katarzyna (Kasia) Siudeja, chercheuse en génomique
Katarzyna (Kasia) Siudeja, CRCN Inserm researcher, has joined the Institute for Integrative Biology of the Cell (I2BC) in Gif sur Yvette in January 2023. She is leading a team Nuclear Dynamics and Repetitive DNA in Tissue Homeostasis. After obtaining her Master’s degree in biotechnology from the University of Wrocław, Poland, Kasia has started her scientific “voyage” through Europe. A short-term fellowship in Greece, followed by a PhD in the Netherlands, have introduced her to the use of the fruit fly, Drosophila melanogaster, as a model system to ask fundamental questions in biology. During her PhD, in the laboratory of Ody Sibon in the University Medical Center in Groningen, The Netherlands, she studied mechanisms underlying a rare neurodegenerative disease caused by genetic mutations in the Coenzyme A biosynthesis pathway. For her postdoctoral training, Kasia moved to the team of Allison Bardin in the Institut Curie in Paris, to study how and why somatic cells acquire DNA mutations, and how these mutations can impact cell function. Using the Drosophila intestine, a simplified but powerful model of a self-renewing tissue, she demonstrated that aging fly adult stem cells accumulate mutations through mechanisms similar to those operating in human cancers. Recruited as an Inserm researcher in 2016, she continued to develop genetic and genomic tools to better understand somatic genetic variation in the fly gut. This work has later uncovered prevalent somatic activity of transposable elements (TEs) in the fly intestine. TEs are omnipresent repetitive DNA sequences, some of which are able to mobilize and propagate in the host genome. TE activity is proposed to contribute to aging, cancer and other pathologies, however their mechanisms of function in somatic tissues are not well understood. Thanks to an ERC Starting grant obtained in 2022, Kasia and her team are now addressing how TEs and other repetitive DNA sequences influence cell and tissue function, including their impact on stem cel function and tissue aging. “Nothing in life is to be feared, it is only to be understood.” - Maria Skłodowska-Curie Contact : katarzyna.siudeja@i2bc.paris-saclay.fr
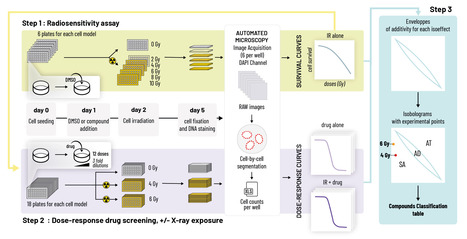
L’efficacité de la radiothérapie sur certaines tumeurs est encore insuffisante et ses effets secondaires parfois invalidants. Les agents dits radiosensibilisants et couramment utilisés en clinique sont peu nombreux, d’une efficacité démontrée mais limitée et non dénués d’effets secondaires car radiosensibilisant également les tissus sains. Dans une étude parue dans International Journal of Radiation Oncology, Biology, Physics (IJROBP) une équipe de l’Unité Chimie et Modélisation pour la Biologie du Cancer (CNRS UMR 9187, Inserm UMR-S 1196, UPSaclay et Institut Curie d’Orsay) a participé au développement d’une approche novatrice pour accélérer la découverte de molécules radiosensibilisant les tumeurs. Cette stratégie s’est appuyée sur la méthodologie de criblage à haut-débit de la plateforme BioPhenics (Institut Curie) en y intégrant un dispositif d’irradiation et une analyse informatique automatisée des interactions de chaque molécule avec les rayons, basée sur la méthode de référence dite isobolographique. L’étude menée en collaboration entre radio-oncologue, radiobiologiste, ingénieurs biologistes, chimistes, physicien et bio-informaticien a permis de tester avec succès une collection de 160 molécules candidates (issues principalement de la chimiothèque de l’Institut Curie) directement sur des cellules tumorales de glioblastome, de cancer du col de l’utérus et sur des fibroblastes normaux. Parmi les 15 composés identifiés comme radiosensibilisant très significativement les cellules tumorales, 5 affichent une faible toxicité sur les fibroblastes normaux. Cette stratégie innovante à haut-débit constitue désormais un outil précieux pour identifier rapidement des composés capables de majorer significativement l’efficacité de la radiothérapie sans en augmenter la toxicité (majoration de l’index thérapeutique) mais également des composés radioprotecteurs. Contact : pierre.verrelle@curie.fr
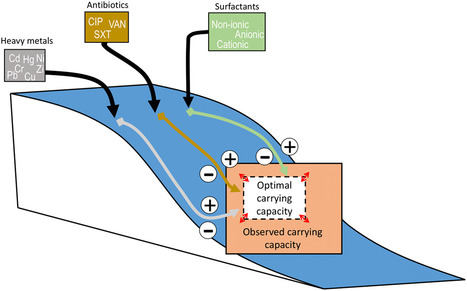
L'émergence et la sélection de la résistance aux antibiotiques constituent un problème majeur de santé publique. La présence de bactéries résistantes aux antibiotiques dans les environnements naturels et anthropisés menace la durabilité des efforts visant à réduire la résistance dans les populations humaines et animales. Dans un article publié dans Science of the Total Environment, des chercheurs du Laboratoire Epidémiologie et modélisation de la résistance aux antimicrobiens (CESP, Institut Pasteur, Université Paris-Saclay), en collaboration avec des chercheurs de l’Université de Limoges ont développé un modèle mathématique permettant d’explorer l’impact de la présence de certains polluants dans les eaux usées (antibiotiques, métaux lourds, tensioactifs) sur la dynamique de résistance aux antibiotiques. En analysant des données récoltées dans des eaux usées urbaines et hospitalières entre 2012 et 2015, ils ont notamment identifié la vancomycine et le mercure comme de potentiels importants co-sélecteurs de certains gènes de résistance aux antibiotiques. Contact : paul.henriot@protonmail.com
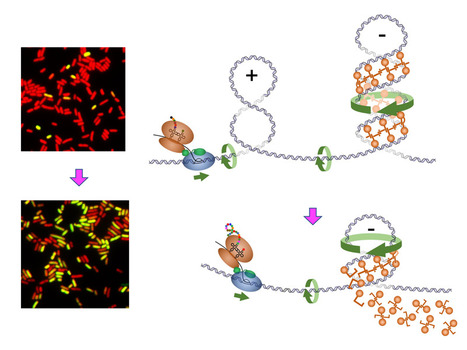
L’ADN du chromosome bactérien est compacté plus de 1000 fois à l'intérieur de la bactérie. Cette condensation est rendue possible par un léger déroulement de la double hélice (surenroulement négatif) entraînant la formation de boucles (plectonèmes), ainsi que par l’interaction de protéines qui compactent davantage ces boucles. Cependant, ces protéines structurales de la chromatine bactérienne entravent le processus de transcription et doivent être temporairement délogées pour permettre l’expression des gènes. C'est le cas de H-NS, une protéine abondante qui bloque l’expression de nombreux gènes. Chez les bactéries pathogènes à Gram négatif, telles que Salmonella, H-NS réprime l’expression de la plupart des gènes de virulence. L’expression de ces gènes au cours de l’infection nécessite donc le déplacement de H-NS. Dans une étude publiée dans Nature Communications, des chercheurs du département de Biologie des Génomes de l'l2BC (CNRS/CEA/UPSaclay, Gif-sur-Yvette) montrent que la fixation de H-NS à l’ADN est déstabilisée par l’accumulation de supertours positifs en amont du complexe d’élongation de la transcription. Ce surenroulement positif résulte de la rotation de l’axe de l’ADN pendant la transcription. Afin d’expliquer la déstabilisation du complexe H-NS:ADN par le surenroulement positif, les chercheurs proposent un modèle dans lequel les oligomères de H-NS joueraient le rôle d’un échafaudage dans l’ADN surenroulé négativement (en faisant le pont entre les bras opposés du plectonème). L’accumulation de supertours positifs et la rotation axiale de l’ADN lors de l’arrivée de l’ARN polymérase provoqueraient l’écroulement de cet échafaudage, suite au déroulement du plectonème, permettant ainsi à l’ARN polymérase de poursuivre son chemin. Contact : lionello.bossi@i2bc.paris-saclay.fr
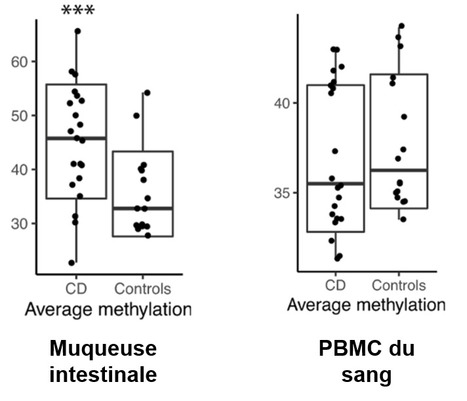
La méthylation de certains sites du génome peut contribuer à la causalité des maladies multifactorielles. La méthylation peut aussi être altérée par les maladies, sans, le plus souvent, qu’on puisse choisir entre les deux possibilités. Dans une étude parue dans Clinical Epigenetics, des chirurgiens de l’hôpital Bicêtre ont étudié, avec l’équipe Inserm d’épigénétique de Pierre Bougnères (MIRCen, Institut de biologie François Jacob, CEA de Fontenay-aux-Roses), la méthylation de la région régulatrice du gène CDH1, qui code pour une protéine de la barrière intestinale, la e-cadhérine. Ils ont analysé ce locus dans des prélèvements chirurgicaux de muqueuse iléale enflammée, atteinte typique de la maladie de Crohn. Ils y ont mis en évidence une hyperméthylation du locus CDH1, comparée à la muqueuse de contrôles. Cette hyperméthylation n’est pas retrouvée dans les cellules du sang. S’agit-il d’un facteur digestif de prédisposition à la maladie, postérieur à la différenciation embryonnaire, ou d’une conséquence de l’inflammation iléale ? Il ne sera pas facile de trancher. Primitive ou secondaire, l’hyperméthylation de ce locus est suspectée de réduire l’expression de CDH1, un gène onco-suppresseur, et de favoriser ainsi le cancer colo-rectal, auquel la maladie de Crohn prédispose. Ceci pourrait faire de la méthylation iléale de CDH1 un biomarqueur précieux de risque de cancer. Les chirurgiens de Bicêtre auteurs de la publication (Solafah Abdalla, Antoine Brouquet, Christophe Penna), avec la collaboration du service de gastroentérologie, ont désormais les cartes en main pour étudier cette hypothèse avec la collaboration des gastroentérologues. Légende Figure : La méthylation est fortement augmentée dans la muqueuse inflammatoire de la maladie de Crohn, pas du tout dans les cellules sanguines, révélant une anomalie épigénétique locale. La muqueuse des contrôles a le même niveau de méthylation que les cellules sanguines. Contact : pierre.bougneres@inserm.fr
|
La Fédération d’Hématologie de l’Université Paris-Saclay, créée en 2022, regroupe les équipes d'hématologie expérimentale du CEA de Fontenay aux Roses, de Gustave Roussy à Villejuif, des hôpitaux de Bicêtre et Paul Brousse et de l'institut Curie à Orsay. Elle organise pour la première fois une école d’été du 1er au 3 juillet 2024. Des cours théoriques portant sur l’hématopoïèse normale et pathologique seront dispensés par des chercheurs/médecins/biologistes affiliés à l’UPS durant 2 jours. La 3ème journée sera consacrée à des travaux pratiques qui seront réalisés dans les laboratoires de Gustave Roussy et du CEA de Fontenay aux roses. Par cette école d’été, nous souhaitons offrir à des étudiants de Licence 3 ou Master 1 de l’UPSaclay la possibilité de découvrir/approfondir leurs connaissances en hématologie. Il s’agit d’une opportunité unique d’échanger avec des experts internationalement reconnus en recherche fondamentale ou translationnelle à travers un format qui facilitera les échanges. Grâce à cette formation, les étudiants pourront mieux identifier les thématiques ainsi que les interlocuteurs utiles pour la poursuite de leurs études dans le domaine de l’hématologie au sein de l’UPSaclay. Pour candidater, veuillez compléter le formulaire en ligne avant le 3 mai 2024. Si vous souhaitez plus de détails, n’hésitez pas à scanner le QR code sur le flyer de présentation ci-dessus, à vous rendre sur le site de la fédération ou à nous contacter à l’adresse mail suivante : emilie.elvira-matelot@inserm.fr Le Comité d’organisation
La Graduate School Métiers de la Recherche et de l’Enseignement Supérieur (GS MRES) vous a déjà annoncé sa troisième édition du congrès junior pluridisciplinaire destiné aux étudiantes et aux étudiants de l’ensemble de l’Université Paris-Saclay (composantes, établissements-composantes et universités associées) et de toutes les disciplines. Le but est de venir découvrir la diversité des disciplines et des formations rassemblées dans l’Université Paris-Saclay à travers la présentation des travaux de recherche auxquels les étudiantes et les étudiants prennent part au sein des formations. Congrès Junior Pluridisciplinaire jeudi 23 mai 2024 après-midi (et soir) à l’ENS Paris-Saclay Tout comme les 2 années précédentes, les étudiantes et les étudiants ont été nombreux à demander une participation au congrès. Le comité scientifique, composé de membres des différentes Graduate Schools, travaille actuellement à l’étude des demandes et va sélectionner les présentations orales, les posters et les vidéos pré-enregistrées qui seront visibles le jour du congrès. Il devrait y avoir environ 50 présentations orales de 5 minutes, 10 vidéos pré-enregistrées de 5 minutes et 30 posters. L’ensemble des composantes, établissements-composantes et universités associées de l’Université Paris-Saclay, l’ensemble des Graduate Schools, ainsi qu’un grand nombre de disciplines seront fièrement représentées le jour du congrès. Le déroulé de la demi-journée et le programme avec tous les intervenants seront bientôt présents sur le site du congrès. Pour venir assister aux présentations, il faut compléter le « formulaire d’inscription en tant que spectateur / spectatrice » accessible sur le site du congrès (ne pas compléter le « formulaire de demande de participation ») : Les inscriptions fermeront le 26 avril 2024, ne tardez pas ! Que vous soyez étudiant participant ou non, chercheur, enseignant, enseignant-chercheur, administratif, famille ou autre, nous espérons avoir le plaisir de vous compter parmi nous le 23 mai 2024 pour soutenir les étudiantes et étudiants qui présentent et pour découvrir l’ensemble de leurs travaux. Le comité d’organisation du congrès junior de la GS MRES
SAVE THE DATE ! RIC Réseau d'Imagerie Cellulaire Paris-Saclay - Demi-journée Super résolution - 3 octobre 2024
Demi-journée consacrée à l'imagerie super résolutive organisée par le Réseau d'Imagerie Cellulaire de Paris-Saclay et qui aura lieu le 3 octobre 2024 dans l'auditorium du bâtiment recherche de la Faculté de Médecine UPSaclay au Kremlin Bicêtre.
It is our great pleasure to announce the 7th edition of the « Physics and Biological Systems » biennal conference PhysBio2024 taking place on the 26-28th of June 2024 at Ecole Polytechnique, Palaiseau.
As usual, this conference aims to bring together a broad range of physical and life scientists working at the interface between the two disciplines around in-depth talks by first-rate international speakers.
The registration is open HERE at a discount price (early registration) before the 24th of May 2024, and regular price after.
The number of attendees will be limited to 200.
We hope to see you there !
The organizing committee.
Le programme Projets de recherche internationaux de l'Inserm permet de consolider des collaborations avec des partenaires étrangers. Il finance des projets de recherche à hauteur de 12 000 à 15 000 € par an sur 5 ans.
La Graduate School Health & Drug Sciences lance son appel à projet "New collaborations". Il vise à soutenir et à promouvoir la recherche en innovation thérapeutique en initiant de nouveaux projets collaboratifs entre au moins 2 équipes de recherche de la GS (hors plateforme).
Le plateau de phénotypage in vivo et transparisation organise sa 2ème journée d’animation scientifique le 12 juin 2024 à l'INRAE de Jouy-en-Josas. À cette occasion, les scientifiques utilisateurs du plateau présenteront les projets menés au sein du plateau pour illustrer leurs diversités ainsi que les expertises et techniques de phénotypage utilisées pour leurs applications. Cette journée a pour vocation de promouvoir les échanges entre utilisateurs et au-delà puisque l’événement est ouvert à tous. Nous vous présenterons également les activités de R&D conduites cette année et les projets en cours. L'inscription est gratuite mais obligatoire en suivant ce lien (il vous suffit de cliquer sur "inscription" dans le menu de gauche puis de vous connecter avec votre compte HAL ou Sciencesconf. Si vous n'en possédez pas alors il faut en créer un en cliquant sur "je n'ai pas de compte").
Symposium international IJPB 2024 "Modélisation des plantes : Opportunités et défis" organisé par de l’Institut Jean-Pierre Bourgin – IJPB à Versailles du 13 au 15 mai 2024 Le symposium IJPB 2024 comportera trois conférences plénières et quatre sessions thématiques traitant de la modélisation des plantes à différentes échelles, du niveau intracellulaire au niveau macroscopique. Le symposium comprendra également des ateliers et une présentation des infrastructures de l'IJPB, en collaboration avec la plateforme d'imagerie et de microscopie de l'Observatoire des plantes et les équipes "Modélisation et imagerie numérique" et "Paroi cellulaire primaire" de l'IJPB. Les présentations seront données par des orateurs invités et par des participants sélectionnés sur la base des résumés soumis. Ce symposium sera une occasion unique d'évaluer les avancées récentes en matière de modélisation, d'explorer la manière dont elle peut être intégrée expérimentalement et d'examiner comment elle s'adapte à l'augmentation constante de la puissance de calcul et à l'explosion des données. Les inscriptions sont ouvertes. Site internet de la manifestation
Via Life Sciences UPSaclay
La ministre de l’Enseignement supérieur et de la Recherche a annoncé ce 22 avril les noms des 22 scientifiques qui se voient allouer des moyens financiers conséquents pour mener leurs travaux en biologie et en santé. Parmi ceux-ci figure Florent Ginhoux. Florent Ginhoux est diplômé en biochimie de l’université Pierre-et-Marie-Curie (UPMC), Paris-VI. Il a obtenu une maitrise en immunologie a l’Institut Pasteur en 2000 et son doctorat en 2004 a l’UPMC, Paris-VI. En tant que postdoctorant, il a rejoint le laboratoire de Miriam Merad à la Mount Sinai School of Medicine (MSSM) à New York, où il a étudié l’ontogenèse et l’homéostasie des cellules myéloïdes, avec un accent particulier sur les cellules de Langerhans et la microglie. En 2008, il est devenu professeur adjoint au département de Médecine génétique et cellulaire du MSSM et membre de l’Institut d’immunologie du MSSM. Il a rejoint le Singapore Immunology Network (SIgN), A*STAR en mai 2009 en tant que chercheur principal junior puis il est devenu chercheur principal senior en 2014. Il a également rejoint le programme EMBO Young Investigator (YIP) en 2013. Florent Ginhoux est un chercheur « hautement cité » sur le Web of Science depuis 2016. Il est également professeur adjoint invité à l’Institut d’immunologie de Shanghai (Université Jiao Tong) depuis 2015 et professeur adjoint associé à l’Institut d’immunologie translationnelle, SingHealth et Duke NUS à Singapour depuis 2016. Il est actuellement directeur de laboratoire à l’Institut Gustave-Roussy où il se concentre sur les cancers pédiatriques et le rôle des cellules myéloïdes dans la progression tumorale. Il est devenu membre de l’EMBO en 2022. Projet scientifique : MATCH “Mapping microglia – tumor crosstalk to cure pediatric brain cancer” / MATCH « Cartographier l’inter-communication entre la microglie et les cellules tumorales pour guérir le cancer du cerveau chez l’enfant » Les tumeurs sont des écosystèmes complexes où, outre les cellules malignes coexistent de multiples populations stromales et immunitaires (y compris les sous-ensembles lymphoïdes et myéloïdes). Malgré les récents succès cliniques des immunothérapies ayant mis en lumière le rôle crucial joué par les lymphocytes infiltrant la tumeur, certains patients ne répondent pas à ces traitements. Les cellules immunitaires innées sont fortement représentées dans les tumeurs, notamment les macrophages associés aux tumeurs (TAM), qui constituent l’une des principales populations immunitaires. Les TAM représentent des cibles cellulaires pertinentes pour concevoir de nouvelles immunothérapies. Cependant, la plupart des stratégies actuelles visant à les cibler par reprogrammation, inhibition du recrutement ou destruction directe ne tiennent pas compte de leur hétérogénéité. Le projet MATCH propose de mieux caractériser les TAM des gliomes pédiatriques du tronc cérébral (GITC) et de développer des modèles précliniques innovants bases sur des organoïdes de cerveaux générés à partir de cellules souches pluripotentes induites du patient. Ces organoïdes de cerveaux contiendront en plus les cellules cancéreuses et les TAM du même patient, permettant une étude compréhensive de leurs interactions afin de développer de nouvelles stratégies thérapeutiques.
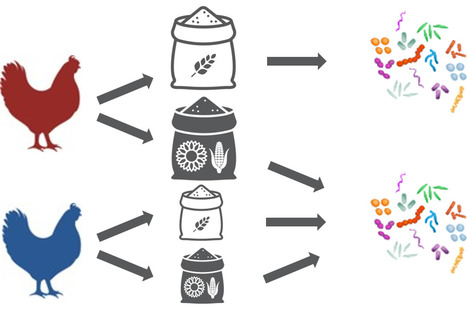
L'alimentation des poules pondeuses représente 60 à 70% des coûts de production, rendant l'efficience alimentaire cruciale pour des raisons économiques et écologiques. Une poule efficiente valorisera mieux les aliments et réduira donc son impact sur l’environnement. Les études indiquent que l'efficience est partiellement déterminée par la génétique, mais le microbiote intestinal, en participant à la dégradation des aliments non digérés par l'animal, pourrait également jouer un rôle. Dans une étude publiée dans Scientific Reports, les scientifiques de l’unité de Génétique Animale et Biologie Intégrative – GABI (INRAE/AgroParisTech/UPSaclay, Jouy-en-Josas) ont examiné le microbiote cæcal de deux lignées de poules pondeuses, sélectionnées de manière divergente depuis plus de 40 ans sur leur efficience alimentaire, et nourries avec deux régimes alimentaires distincts, l'un, CTR, riche en amidon et l'autre, LE, appauvri en énergie et riche en fibres, composant ainsi 4 groupes d’étude. Quel que soit le régime, la lignée efficiente mange en moyenne 2 fois moins que la lignée non efficiente pour une même croissance et une même production d’œufs. Les résultats révèlent que le régime alimentaire affecte significativement la composition du microbiote intestinal des poules pondeuses, mais cette influence varie selon la lignée. Les microbiotes des deux lignées divergent sous le régime CTR, mais se ressemblent avec le régime LE, reflétant in fine deux écosystèmes distincts malgré les 4 conditions étudiées. Le premier, chez les poules moins efficientes nourries avec le régime CTR, est caractérisé par la présence accrue de bactéries fermentant l'amidon. Le second, chez les poules efficientes quel que soit le régime et celles moins efficientes nourries avec le régime LE, inclut des bactéries fibrolytiques produisant du propionate. Ce dernier traverse l'intestin et stimule la production de glucose dans le foie. Il pourrait ainsi contribuer à améliorer l'efficience alimentaire observée sous le régime CTR. Contact : maria.bernard@inrae.fr ou tatiana.zerjal@inrae.fr
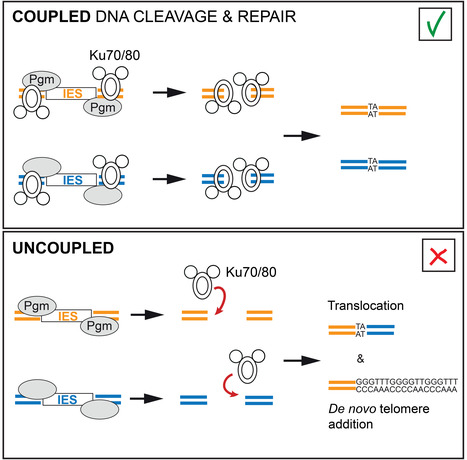
Pas de Ku - pas de coupure : comment la paramécie se protège contre les translocations et la fragmentation de ses chromosomes lorsqu'elle réarrange son génome pendant le développement. Les cassures double-brin de l'ADN (CDB) représentent une menace pour l'intégrité du génome car elles peuvent provoquer des remaniements chromosomiques, causes de maladies ou de mort cellulaire. Pourtant, des processus physiologiques, comme l'assemblage des gènes d'anticorps ou la recombinaison méiotique, reposent sur l'induction de CDB, qui doivent donc être étroitement contrôlées et efficacement réparées. Chez la paramécie, des dizaines de milliers de séquences, appelées IES (Internal Eliminated Sequences), sont excisées du génome somatique à chaque cycle sexuel. Ce processus programmé nécessite l’introduction de CDB aux bornes des IES, puis la réparation précise des sites d’excision par la voie NHEJ de jonction d’extrémités non homologues. La présence d'un variant spécialisé de Ku, un acteur-clé du NHEJ, est nécessaire pour activer les coupures de l’ADN, indiquant l’existence d’un couplage entre cassure et réparation. Dans une étude publiée dans Cell Reports, l’équipe « Réarrangements Programmés du Génome » de l’I2BC (CNRS/CEA/UPSaclay, Gif-sur-Yvette) en collaboration avec l’équipe IntGen, ont montré qu’un mutant de Ku incapable de se fixer à l'ADN active toujours la coupure des bornes des IES, mais est inactif pour leur réparation par le NHEJ. Les cassures non réparées sont dégradées ou servent de substrat pour l’ajout de télomères. En coexprimant les versions sauvage et mutante de Ku, les chercheurs ont découplé les étapes de coupure et de réparation. Par séquençage haut-débit de l’ADN, ils ont détecté une forte augmentation des translocations chromosomiques, démontrant ainsi que le couplage des deux étapes de l’excision garantit la fidélité de l’assemblage du génome pendant les réarrangements programmés. Contact : mireille.betermier@i2bc.paris-saclay.fr ou julien.bischerour@i2bc.paris-saclay.fr
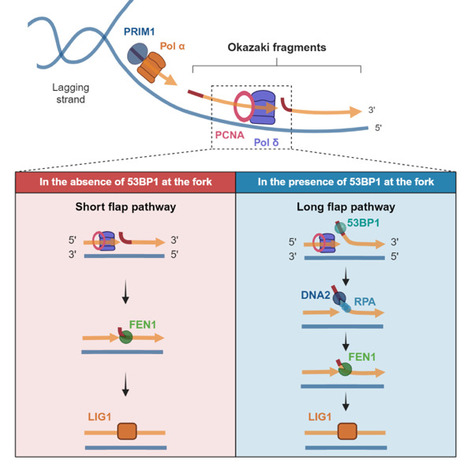
Les protéines de liaison aux ARN sont impliquées dans de nombreux processus cellulaires et notamment le maintien de l’intégrité du génome. 53BP1 est un régulateur central dans ce mécanisme, agissant dans des contextes de stress cellulaire, tels que le stress réplicatif ou les cassures double-brins de l’ADN. Par ailleurs, ses fonctions dans la signalisation de la réponse aux cassures d’ADN, en favorisant la réparation par NHEJ (Non-Homologous End Joining), seraient dépendantes de l’ARN. Dans une étude publiée dans Cell Reports, Mélissa Leriche et Clara Bonnet, de l’équipe de Stéphan Vagner de l’UMR 3348/INSERM U1278 (CNRS/INSERM/UPSaclay/Institut Curie, Orsay), ont démontré in vitro et in vivo que 53BP1 interagit directement avec des chimères ARN-ADN, identifiées comme étant les fragments d’Okazaki, molécules initiatrices de la réplication de l’ADN. De plus, dans des conditions de réplication non stressées, la déplétion de 53BP1 perturbe la synthèse de l’ADN en modifiant le processus de maturation des fragments d’Okazaki. Par conséquent, 53BP1 permettrait de maintenir l’intégrité de l’ADN lors de la réplication et en limitant le taux de mutation. L’impact de 53BP1 dans les processus de réplication doit être analysé en détail, non seulement car des défauts de maturation des fragments d’Okazaki sont sources d’instabilité génétique, et donc de cancers, mais aussi car 53BP1 est impliquée dans la sensibilité des cellules tumorales déficientes en BRCA1 vis-à-vis des médicaments anticancéreux PARPi (inhibiteurs de PARP). Cette recherche ouvre donc de nouvelles perspectives pour mieux comprendre les mécanismes régulant l’intégrité du génome des cellules. Légende Figure : Illustration du rôle de 53BP1 au niveau de la fourche de réplication. En absence de 53BP1 panneau de gauche, la voie de maturation des fragments d’Okazaki privilégiée est la voie du short flap. Mais en présence de 53BP1, panneau de droite, la voie de maturation favorisée est celle du long flap. Cette dernière serait corrélée à une plus grande activité transcriptionnelle de la région génomique considérée. Contact : patricia.uguen@universite-paris-saclay.fr ou stephan.vagner@curie.fr
Portrait Jeune Chercheur - Samir Bensaid, chercheur en physiologie et biologie de l'exercice
Samir Bensaid a tracé un parcours académique centré sur la pluridisciplinarité depuis la fin de son doctorat en 2019. Avec une passion ardente pour la recherche dans le domaine de la santé et de l'exercice, il a consacré ses premières années de doctorat à approfondir sa compréhension des mécanismes sous-jacents à l'atrophie musculaire. Ses travaux novateurs ont ouvert de nouvelles perspectives et champs de travail au sien de l’Unité de Recherche Pluridisciplinaire Sport, Santé, Société (URePSSS), à l’Université de Lille (France). Ses premières expériences cliniques au Canada et en France l'ont profondément marqué et ont jeté les bases de son engagement à trouver des solutions efficaces dans la prise en charge de personnes souffrant de pathologie métabolique. Après l’obtention de son doctorat, Samir a rejoint l'Université de Lille en tant qu'enseignant-chercheur contractuel, où il a rapidement élargi son champ d'expertise. Il a eu l’opportunité de se rapprocher et de collaborer avec des chercheurs de renom, le Pr. Elsa Heyman, le Pr. Frédéric Daussin et le Dr. Semah Tagougui sur plusieurs de leurs projets : un projet fondamental « Optimisation de l’activité physique par une supplémentation en flavanol de cacao chez les sujets diabétiques de type 1 » et un projet clinique « Caractériser les effets de la chirurgie bariatrique et l’impact du niveau d’activité physique ». Puis en 2021, Samir a réalisé un stage postdoctoral dans l’unité de recherche en physiologie moléculaire du Dr. Mathieu Ferron à l’institut de recherches cliniques de Montréal (IRCM) sur un projet fondamental : « Rôle de la vitamine K dans la prévention de la stéatose-hépatique non alcoolique (NASH) ». Depuis 2023, Samir poursuit sa carrière en tant que chercheur en physiologie et biologie de l'exercice au Centre d'Études et de Recherches pour l'Intensification du Traitement du Diabète (CERITD) au Centre Hospitalier Sud Francilien. Il est membre du Laboratoire de biologie de l’exercice pour la performance et la santé (LBEPS) (UPSacay/UEVE/IRBA, Evry). Son engagement dans des projets de recherche clinique sur le diabète, en particulier sur l'impact de l'activité physique, témoigne de sa volonté de traduire des découvertes scientifiques en interventions pratiques bénéfiques pour les patients. Son projet "ACTIDIAB" représente une avancée significative dans la compréhension de l'effet de l'activité physique sur le contrôle glycémique. Samir souhaite démontrer que l'activité physique peut être aussi efficace, voire plus, que certains médicaments pour améliorer le contrôle glycémique et réduire les complications associées à l’insulinorésistance. « La science est la quête incessante du savoir, la recherche inlassable de la vérité. » - Averroès Contact : samir.bensaid@ceritd.com
|






 Your new post is loading...
Your new post is loading...